
Capítulo 11 Análise de Variância para 2 fatores
Também conhecida como 2-way Anova, é utilizada quando um estudo ou experimento é desenhado para responder a mais questões que uma Anova para 1 fator é capaz de responder.
É muito comum comparar tratamentos que são combinações de dois fatores independentes.
A inferência para uma Anova de dois fatores é feita da seguinte forma:
- Faça gráficos das médias dos grupos
- Procure por interação
- Procure por efeitos principais
- Faça o teste F para três questões:
- Há interação?
- Há efeito principal para o primeiro fator?
- Há efeito principal para o segundo fator?
A seguir veremos três exemplos que ilustram várias situações da Anova de 2 fatores.
Exemplo 11.1 (Análise de Variância para dois fatores - interação não significativa) A reprodução tem um custo fisiológico alto. Uma dieta rica em proteínas pode aumentar a capacidade reprodutiva de moscas das frutas.
Um experimento avaliou a porcentagem de gordura corporal em 40 fêmeas de mosca das frutas alimentadas com 4 dietas enriquecidas com proteínas (ou não) nas doses de 0, 1, 3 e 7 g.
O experimento usou moscas selvagens e mutantes com ciclo reprodutivo mais longo.
A resposta foi a porcentagem de gordura corporal após duas semanas da dieta, contidas no arquivo mosca.xlsx.
São três grupos de hipóteses a serem testadas:
H0: não há interação entre os genótipos e quantidade de proteína na porcentagem de gordura corporal de moscas.
H1: há interação entre os genótipos e quantidade de proteína na porcentagem de gordura corporal de moscas.
H0: a quantidade de proteína não influencia na porcentagem de gordura corporal de moscas.
H1:a quantidade de proteína influencia na porcentagem de gordura corporal de moscas.
H0: o genótipo não influencia na porcentagem de gordura corporal de moscas.
H1: o genótipo influencia na porcentagem de gordura corporal de moscas.
Análise exploratória:
mosca <- readxl::read_excel("data/mosca.xlsx") %>%
mutate(
genotipo = factor(genotipo),
proteina = factor(proteina)
)
mosca %>%
group_by(genotipo, proteina) %>%
summarise(
n = n(),
media = mean(gordura),
desvpad = sd(gordura),
var = var(gordura)
)## `summarise()` has grouped output by 'genotipo'. You can override using the
## `.groups` argument.## # A tibble: 8 × 6
## # Groups: genotipo [2]
## genotipo proteina n media desvpad var
## <fct> <fct> <int> <dbl> <dbl> <dbl>
## 1 mutante 0 5 24.2 1.76 3.09
## 2 mutante 1 5 23.3 2.51 6.32
## 3 mutante 3 5 14.6 3.17 10.1
## 4 mutante 7 5 13.0 2.71 7.33
## 5 selvagem 0 5 25.6 1.50 2.24
## 6 selvagem 1 5 22.3 1.72 2.96
## 7 selvagem 3 5 15.1 0.691 0.477
## 8 selvagem 7 5 11.6 1.06 1.13
## Boxplot - Efeito principal fator A
boxplot(gordura ~ genotipo, data = mosca)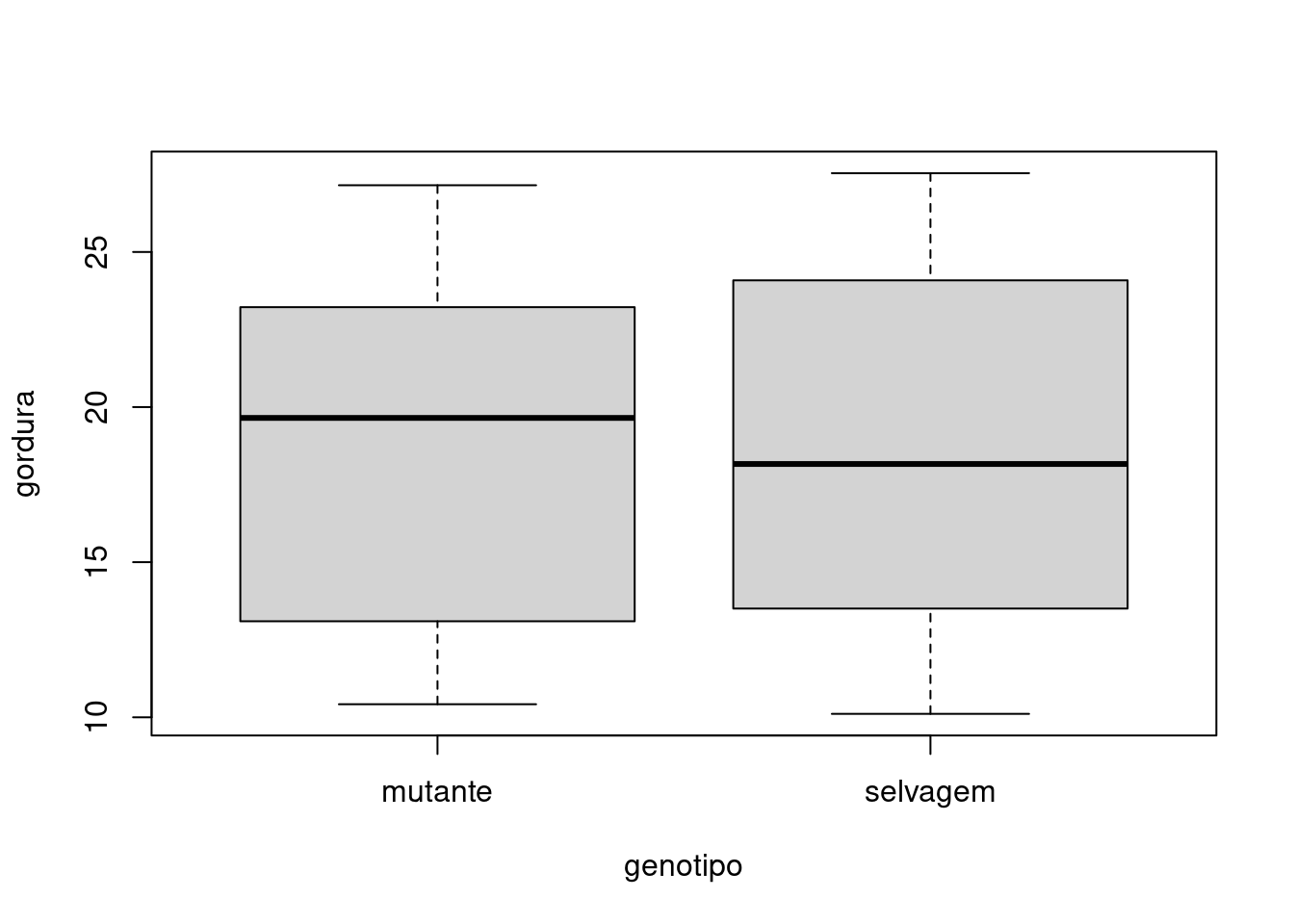
## Boxplot - Efeito principal fator B
boxplot(gordura ~ proteina, data = mosca)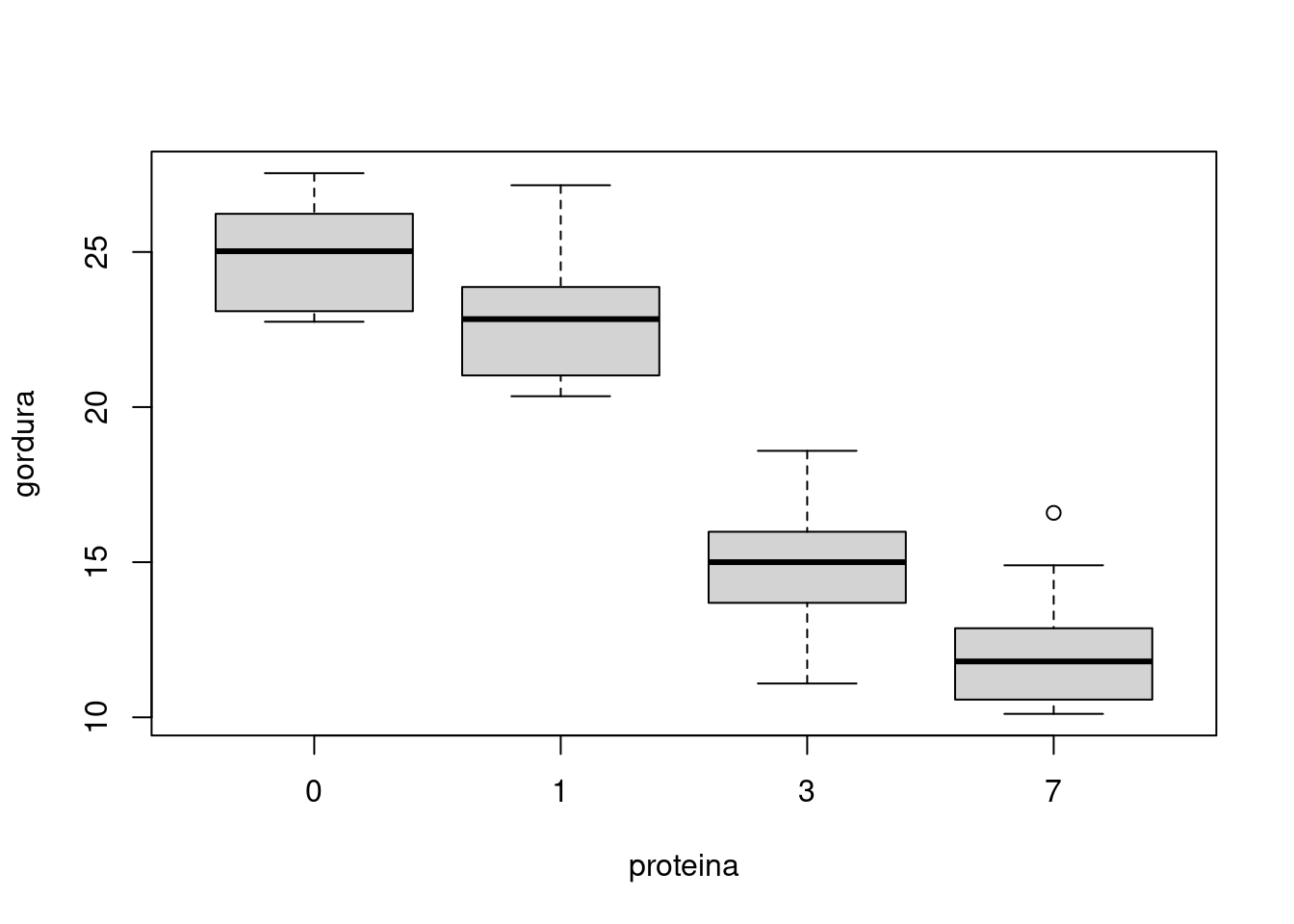
## Boxplot - Interação
boxplot(gordura ~ genotipo * proteina, data = mosca)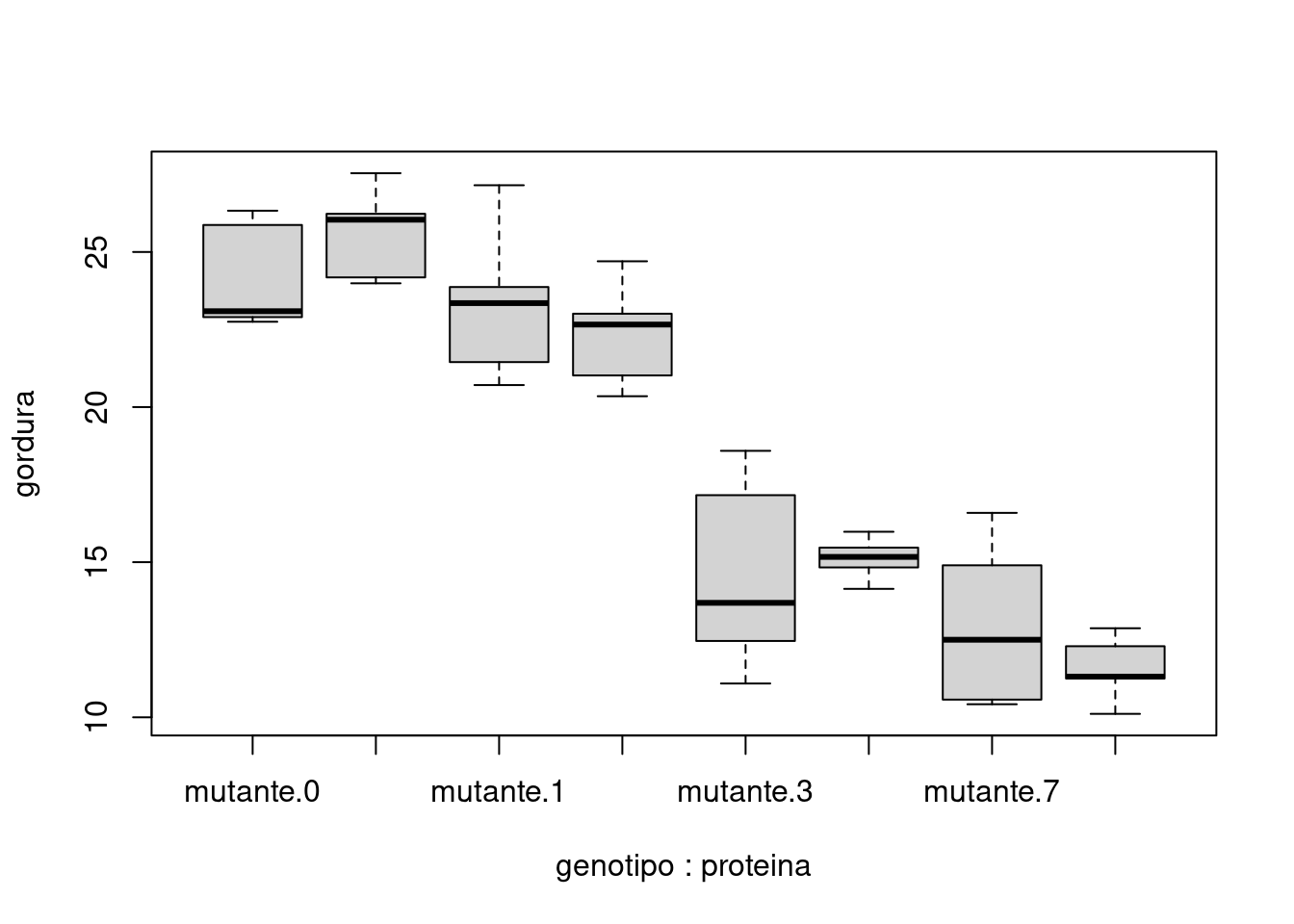
## Gráfico da interação
with(mosca, interaction.plot(genotipo, proteina, gordura))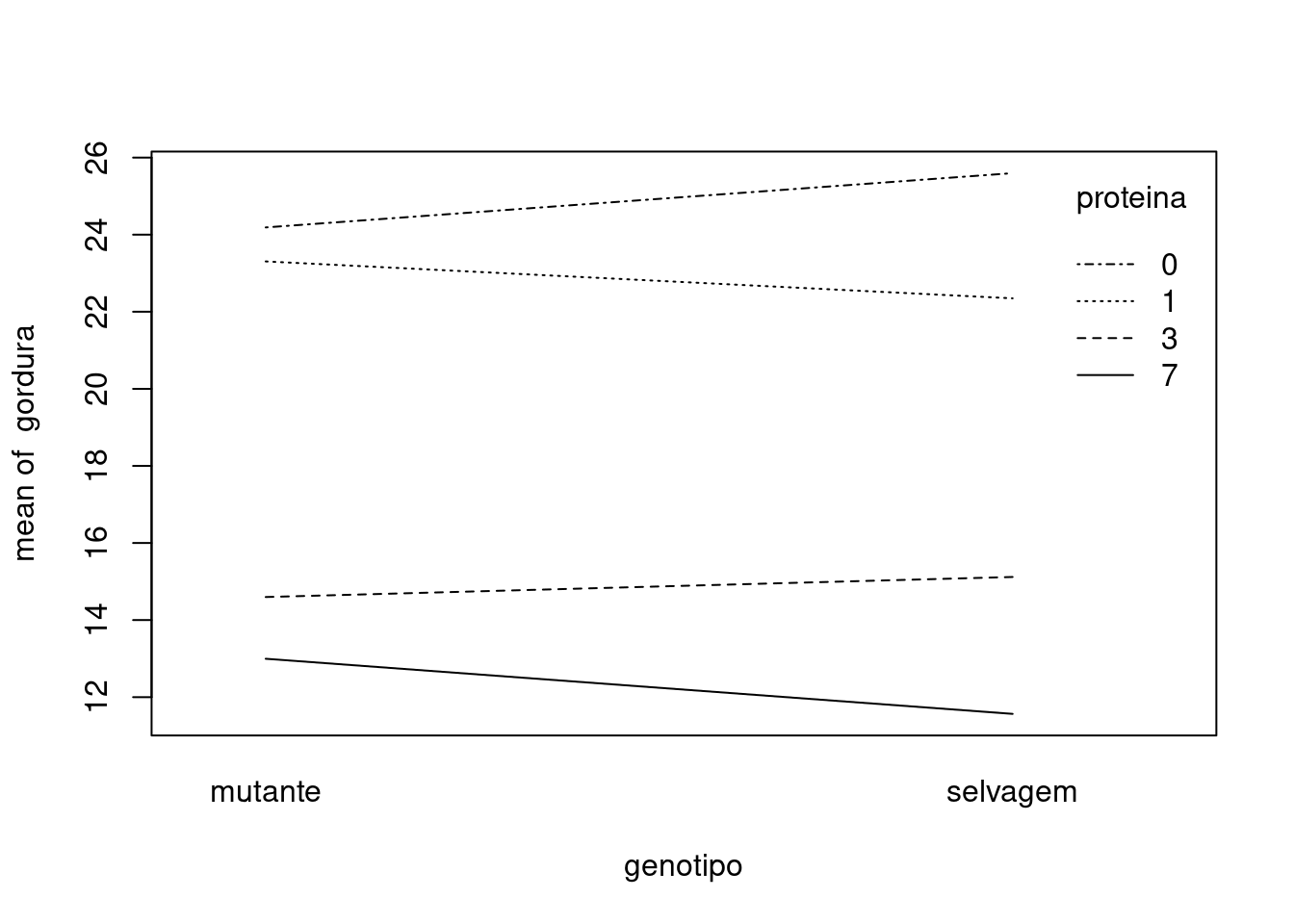
with(mosca, interaction.plot(proteina, genotipo, gordura))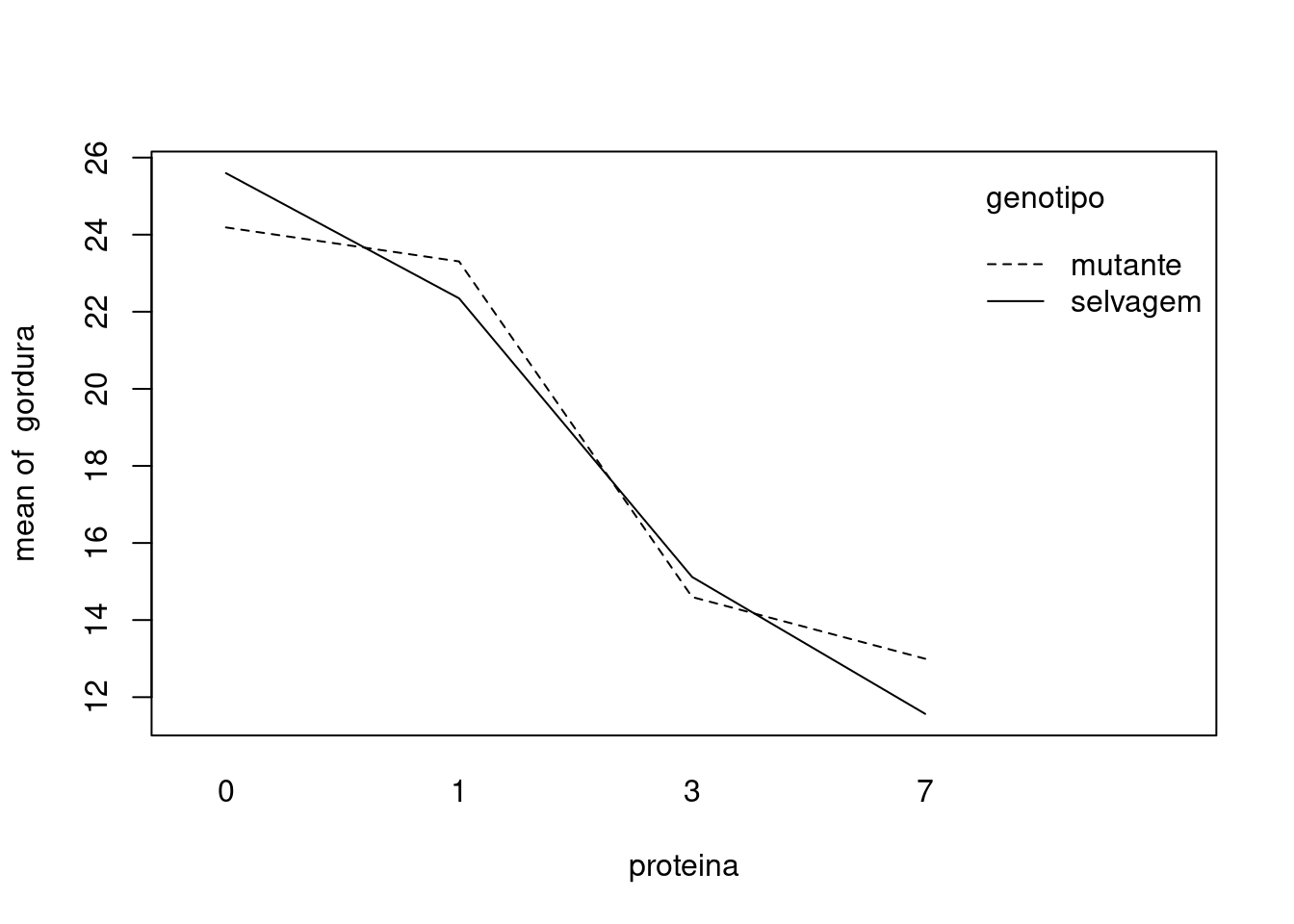
Efetuamos a Análise de Variância para os efeitos principais e para a interação:
## Analysis of Variance Table
##
## Response: gordura
## Df Sum Sq Mean Sq F value Pr(>F)
## genotipo 1 0.13 0.13 0.0315 0.8602
## proteina 3 1113.37 371.12 88.3984 1.429e-15 ***
## genotipo:proteina 3 12.91 4.30 1.0247 0.3947
## Residuals 32 134.35 4.20
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1A interação (p-valor=0,395) e o efeito principal para genótipo (p-valor=0,86) são pequenos (não significativos). O efeito principal para proteína (p-valor < 0,001) é grande (significativo).
Verificação do presuposto da Normalidade.
## [1] 2.495927## [1] 0.3766119
## Teste de Shapiro-Wilk
aov_mosca %>%
residuals() %>%
shapiro.test()##
## Shapiro-Wilk normality test
##
## data: .
## W = 0.97295, p-value = 0.444
## Gráfico dos quantis normais
aov_mosca %>%
residuals() %>%
qqnorm()
aov_mosca %>%
residuals() %>%
qqline()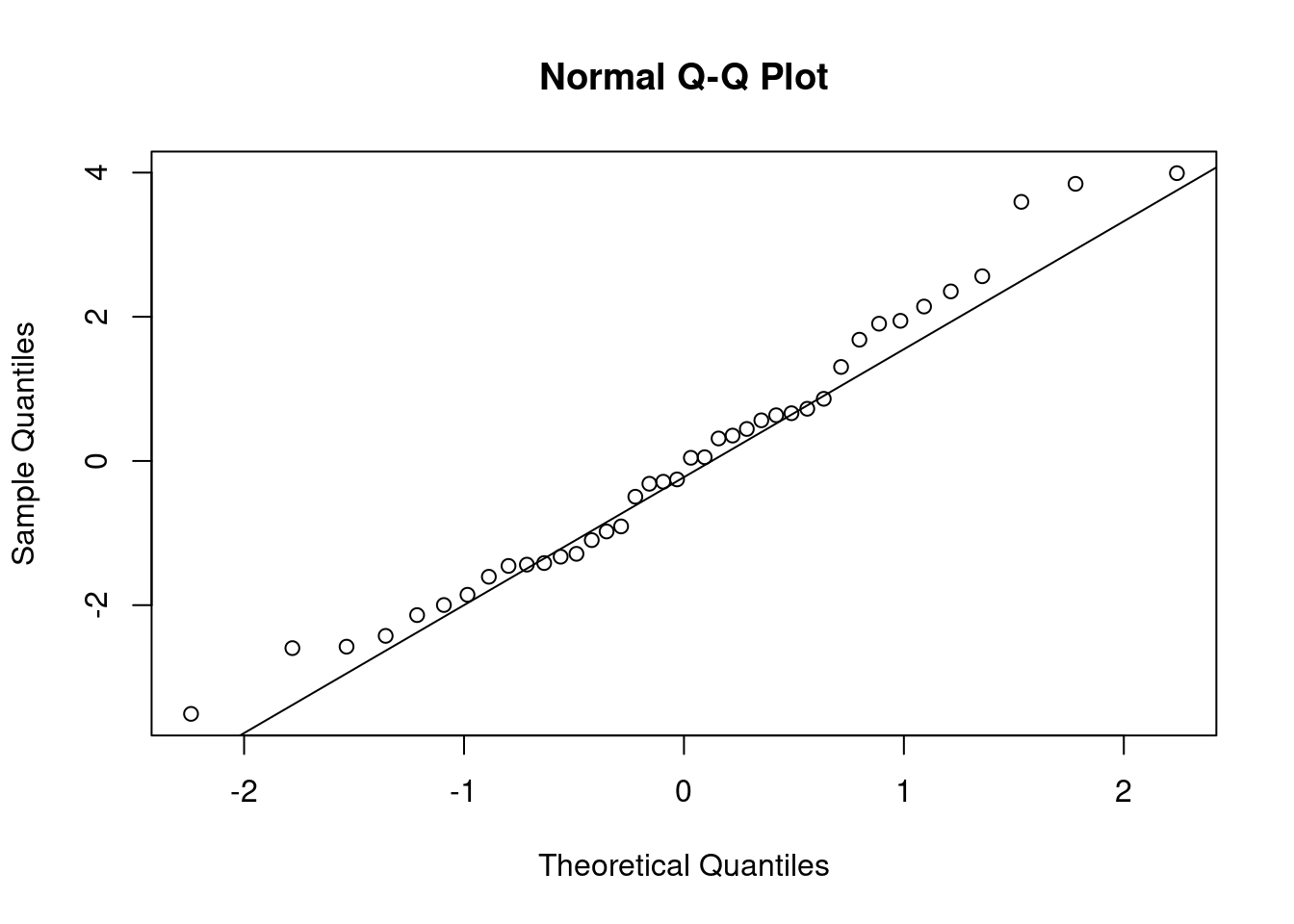
##
## The decimal point is at the |
##
## -3 | 5
## -2 | 66410
## -1 | 965443310
## -0 | 95333
## 0 | 0134466779
## 1 | 3799
## 2 | 146
## 3 | 68
## 4 | 0Verificação do presuposto da homogeneidade das variâncias
## razão maior/menor desvio-padrão
mosca %>%
group_by(genotipo, proteina) %>%
summarise(desvpad = sd(gordura)) %>%
mutate(razao = max(desvpad) / desvpad)## `summarise()` has grouped output by 'genotipo'. You can override using the
## `.groups` argument.## # A tibble: 8 × 4
## # Groups: genotipo [2]
## genotipo proteina desvpad razao
## <fct> <fct> <dbl> <dbl>
## 1 mutante 0 1.76 1.80
## 2 mutante 1 2.51 1.26
## 3 mutante 3 3.17 1
## 4 mutante 7 2.71 1.17
## 5 selvagem 0 1.50 1.15
## 6 selvagem 1 1.72 1
## 7 selvagem 3 0.691 2.49
## 8 selvagem 7 1.06 1.62
## boxplot condicional dos resíduos
boxplot(residuals(aov_mosca) ~ genotipo * proteina, data = mosca)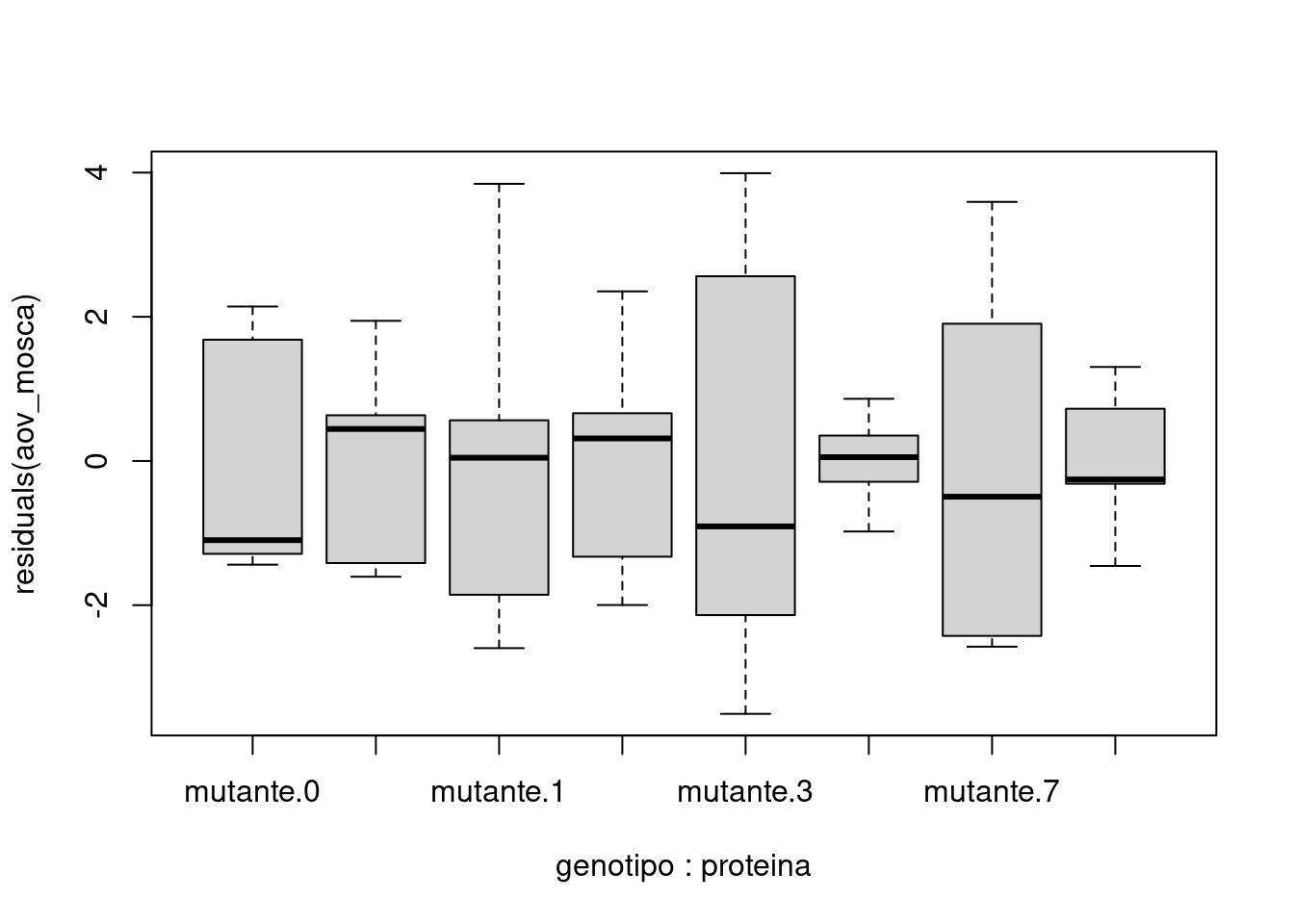
## resíduos vs ajustados
plot(aov_mosca, 1)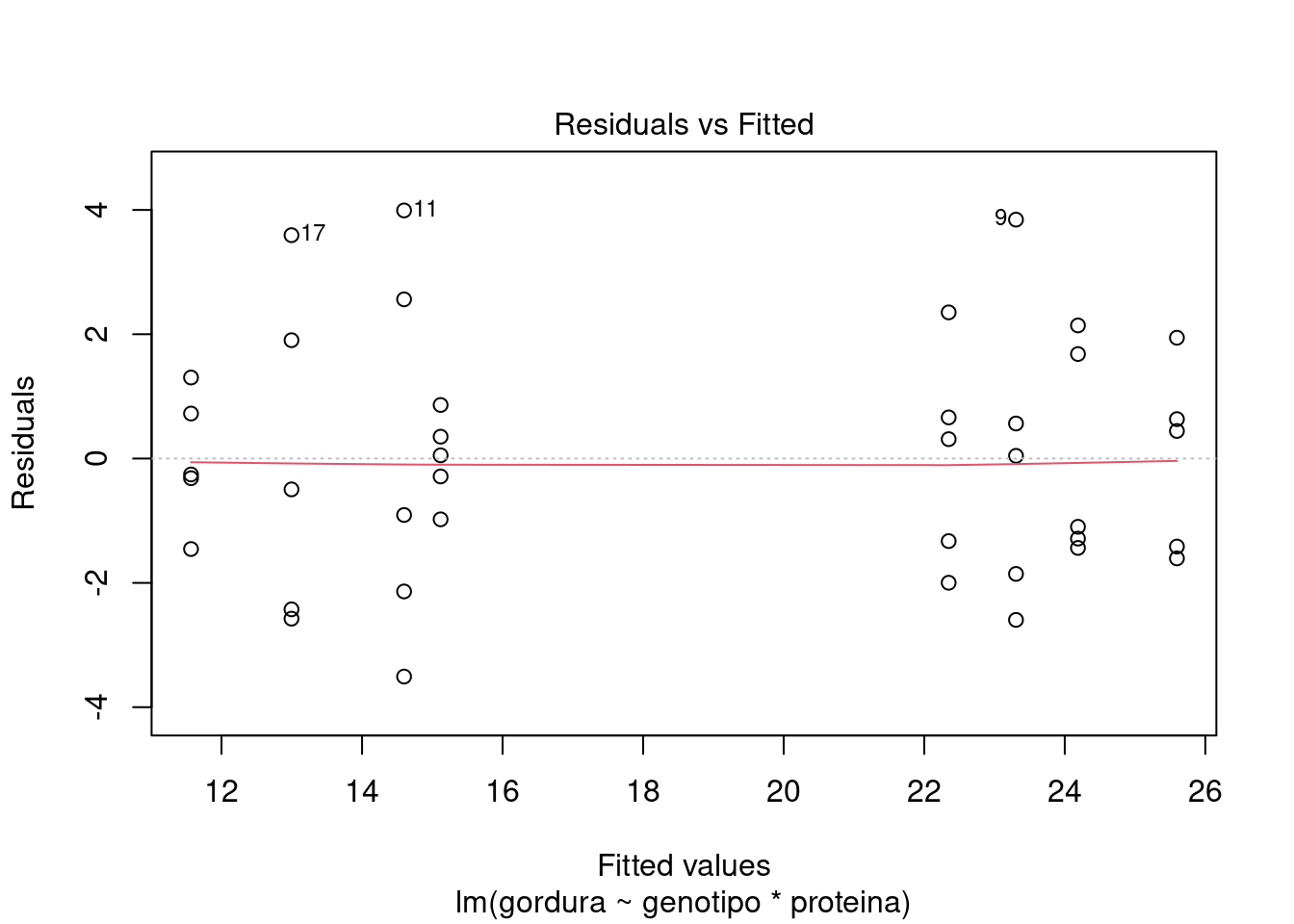
## teste de Bartlett
bartlett.test(residuals(aov_mosca) ~ paste0(genotipo, proteina), data = mosca)##
## Bartlett test of homogeneity of variances
##
## data: residuals(aov_mosca) by paste0(genotipo, proteina)
## Bartlett's K-squared = 10.739, df = 7, p-value = 0.1504
## teste de Levene
car::leveneTest(residuals(aov_mosca) ~ paste0(genotipo, proteina), data = mosca)## Warning in leveneTest.default(y = y, group = group, ...): group coerced to
## factor.## Levene's Test for Homogeneity of Variance (center = median)
## Df F value Pr(>F)
## group 7 1.3106 0.2773
## 32Pela análise do conjunto dos resultados acima, não há evidência de violação dos pressupostos.
Teste de comparação de médias para o efeito principal do fator proteína:
tk_proteina <- emmeans::emmeans(aov_mosca, ~proteina, contr = "tukey")## NOTE: Results may be misleading due to involvement in interactions
## contrastes
tk_proteina$contrasts## contrast estimate SE df t.ratio p.value
## proteina0 - proteina1 2.06 0.916 32 2.254 0.1307
## proteina0 - proteina3 10.03 0.916 32 10.950 <.0001
## proteina0 - proteina7 12.61 0.916 32 13.763 <.0001
## proteina1 - proteina3 7.97 0.916 32 8.697 <.0001
## proteina1 - proteina7 10.55 0.916 32 11.509 <.0001
## proteina3 - proteina7 2.58 0.916 32 2.812 0.0395
##
## Results are averaged over the levels of: genotipo
## P value adjustment: tukey method for comparing a family of 4 estimates## contrast estimate SE df lower.CL upper.CL
## proteina0 - proteina1 2.06 0.916 32 -0.4177 4.55
## proteina0 - proteina3 10.03 0.916 32 7.5513 12.52
## proteina0 - proteina7 12.61 0.916 32 10.1283 15.09
## proteina1 - proteina3 7.97 0.916 32 5.4863 10.45
## proteina1 - proteina7 10.55 0.916 32 8.0633 13.03
## proteina3 - proteina7 2.58 0.916 32 0.0943 5.06
##
## Results are averaged over the levels of: genotipo
## Confidence level used: 0.95
## Conf-level adjustment: tukey method for comparing a family of 4 estimates## proteina emmean SE df lower.CL upper.CL .group
## 7 12.3 0.648 32 11.0 13.6 a
## 3 14.9 0.648 32 13.5 16.2 b
## 1 22.8 0.648 32 21.5 24.1 c
## 0 24.9 0.648 32 23.6 26.2 c
##
## Results are averaged over the levels of: genotipo
## Confidence level used: 0.95
## P value adjustment: tukey method for comparing a family of 4 estimates
## significance level used: alpha = 0.05
## NOTE: If two or more means share the same grouping symbol,
## then we cannot show them to be different.
## But we also did not show them to be the same.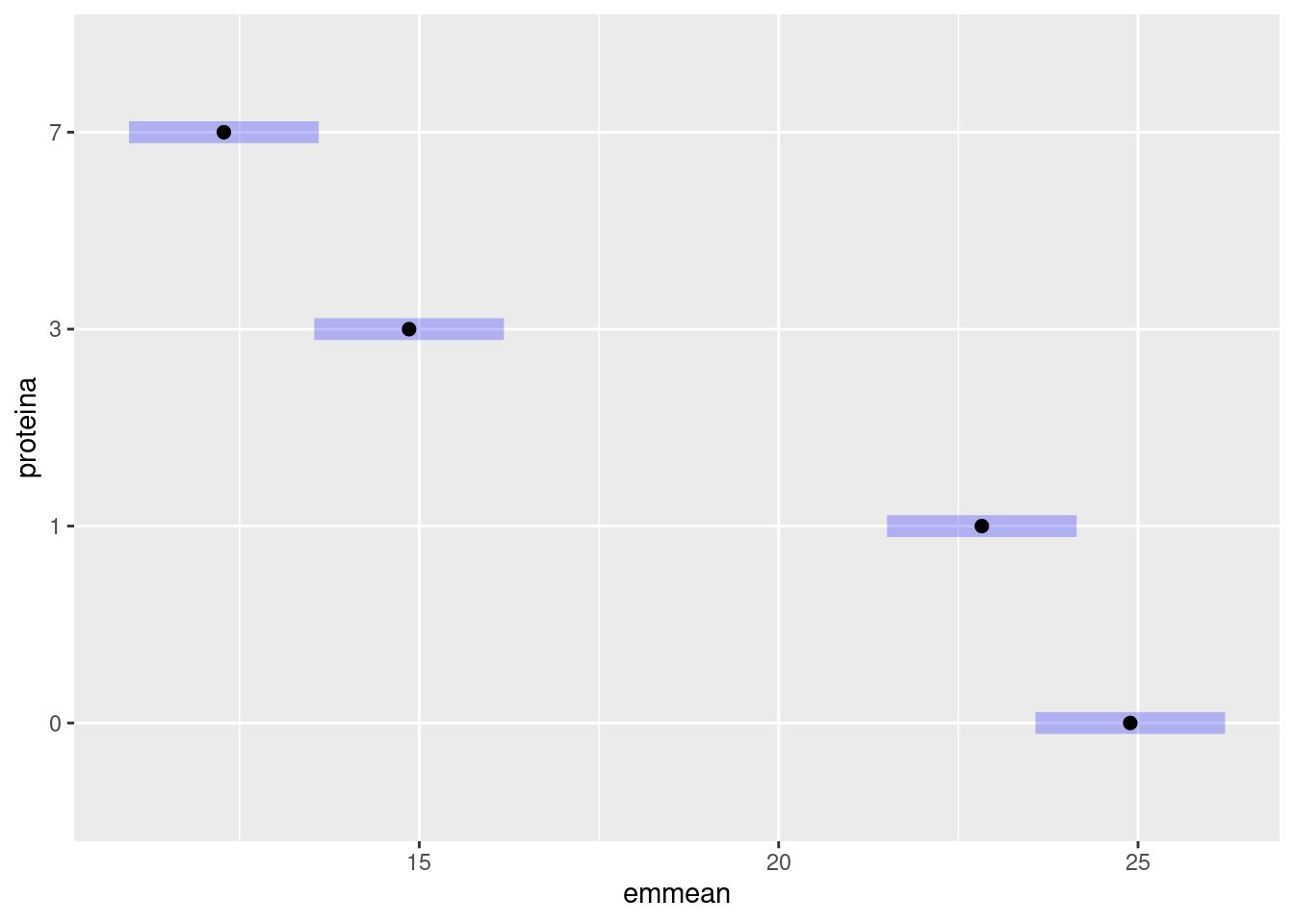
Teste de comparação de médias para o efeito principal do fator genótipo:
tk_genotipo <- emmeans::emmeans(aov_mosca, ~genotipo, contr = "tukey")## NOTE: Results may be misleading due to involvement in interactions
## contrastes
tk_genotipo$contrasts## contrast estimate SE df t.ratio p.value
## mutante - selvagem 0.115 0.648 32 0.177 0.8602
##
## Results are averaged over the levels of: proteina## contrast estimate SE df lower.CL upper.CL
## mutante - selvagem 0.115 0.648 32 -1.2 1.43
##
## Results are averaged over the levels of: proteina
## Confidence level used: 0.95## genotipo emmean SE df lower.CL upper.CL .group
## selvagem 18.7 0.458 32 17.7 19.6 a
## mutante 18.8 0.458 32 17.8 19.7 a
##
## Results are averaged over the levels of: proteina
## Confidence level used: 0.95
## significance level used: alpha = 0.05
## NOTE: If two or more means share the same grouping symbol,
## then we cannot show them to be different.
## But we also did not show them to be the same.
Conclusão: Maiores quantidade de proteína na dieta levam a menores taxas de gordura corporal. O genótipo da mosca das frutas tem um efeito pequeno na gordura corporal para qualquer quantidade de proteína.
O próximo exemplo ilustra uma situação em que a interação é significativa, mas os efeitos principais são grandes e mais importantes.
Exemplo 11.2 (Análise de Variância para dois fatores - interação significativa mas com efeitos principais grandes e mais importantes) Fungos micorrízicos estão presentes nas raízes de muitas plantas. Eles tem uma relação simbiótica com a planta.
Um experimento comparou o efeito da adição de nitrogênio em dois genótipos de tomate, um selvagem suscetível a micorrizas e um mutante não suscetível.
O nitrogênio foi adicionado nas doses de 0, 28 e 10 kg/ha.
Seis plantas foram aleatoriamente designadas a cada grupo.
A variável resposta é a porcentagem de fósforo nas plantas depois de 19 semanas (tomate.xlsx).
São três grupos de hipóteses a serem testadas:
H0 : não há interação entre os genótipos e doses de nitrogênio na porcentagem de fósforo em plantas de tomate.
H1: há interação entre os genótipos e doses de nitrogênio na porcentagem de fósforo em plantas de tomate.
H0: o genótipo não influencia na porcentagem de fósforo em plantas de tomate.
H1: o genótipo influencia na porcentagem de fósforo em plantas de tomate.
H0: as doses de nitrogênio não influenciam na porcentagem de fósforo em plantas de tomate.
H1: as doses de nitrogênio influenciam na porcentagem de fósforo em plantas de tomate.
Análise exploratória:
tomate <- readxl::read_excel("data/tomate.xlsx") %>%
mutate(
genotipo = factor(genotipo),
nitrogenio = factor(nitrogenio)
)
tomate %>%
group_by(genotipo, nitrogenio) %>%
summarise(
n = n(),
media = mean(fosforo),
desvpad = sd(fosforo),
var = var(fosforo)
)## `summarise()` has grouped output by 'genotipo'. You can override using the
## `.groups` argument.## # A tibble: 6 × 6
## # Groups: genotipo [2]
## genotipo nitrogenio n media desvpad var
## <fct> <fct> <int> <dbl> <dbl> <dbl>
## 1 mutante 0 6 0.248 0.0306 0.000937
## 2 mutante 28 6 0.207 0.0242 0.000587
## 3 mutante 160 6 0.182 0.0147 0.000217
## 4 selvagem 0 6 0.512 0.0833 0.00694
## 5 selvagem 28 6 0.423 0.0455 0.00207
## 6 selvagem 160 6 0.318 0.0462 0.00214
## Boxplot - Efeito principal fator A
boxplot(fosforo ~ genotipo, data = tomate)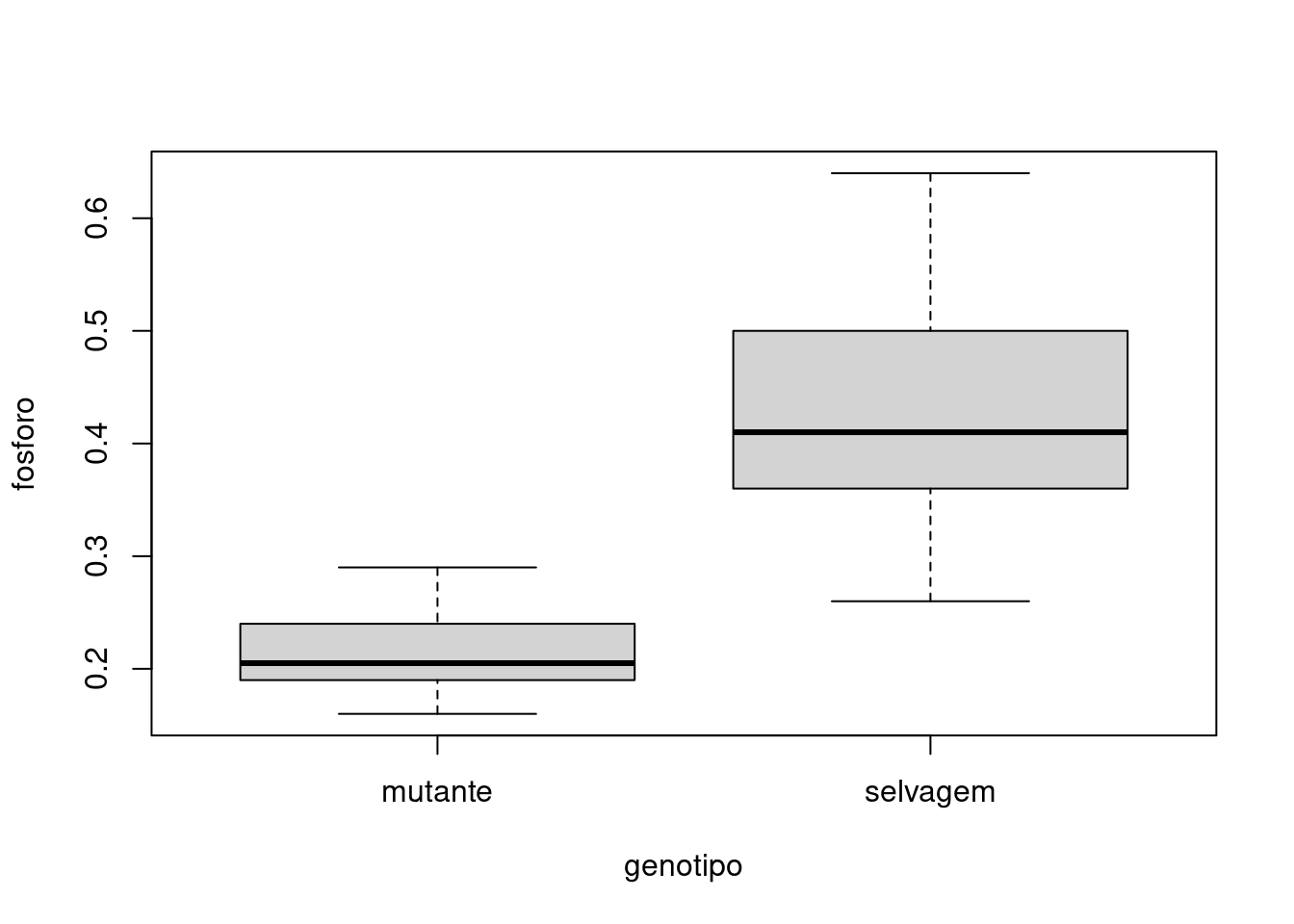
## Boxplot - Efeito principal fator B
boxplot(fosforo ~ nitrogenio, data = tomate)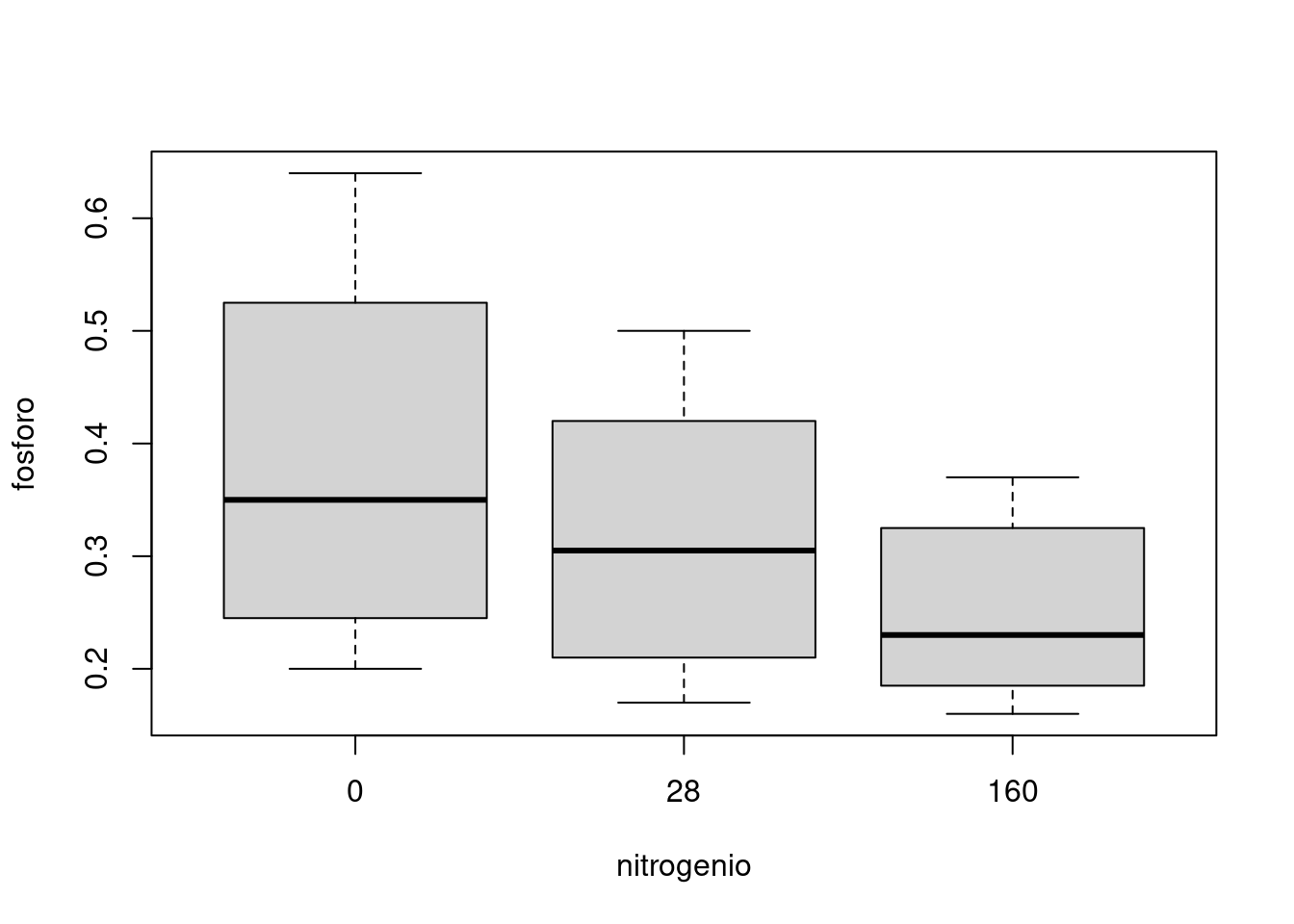
## Boxplot - Interação
boxplot(fosforo ~ genotipo * nitrogenio, data = tomate)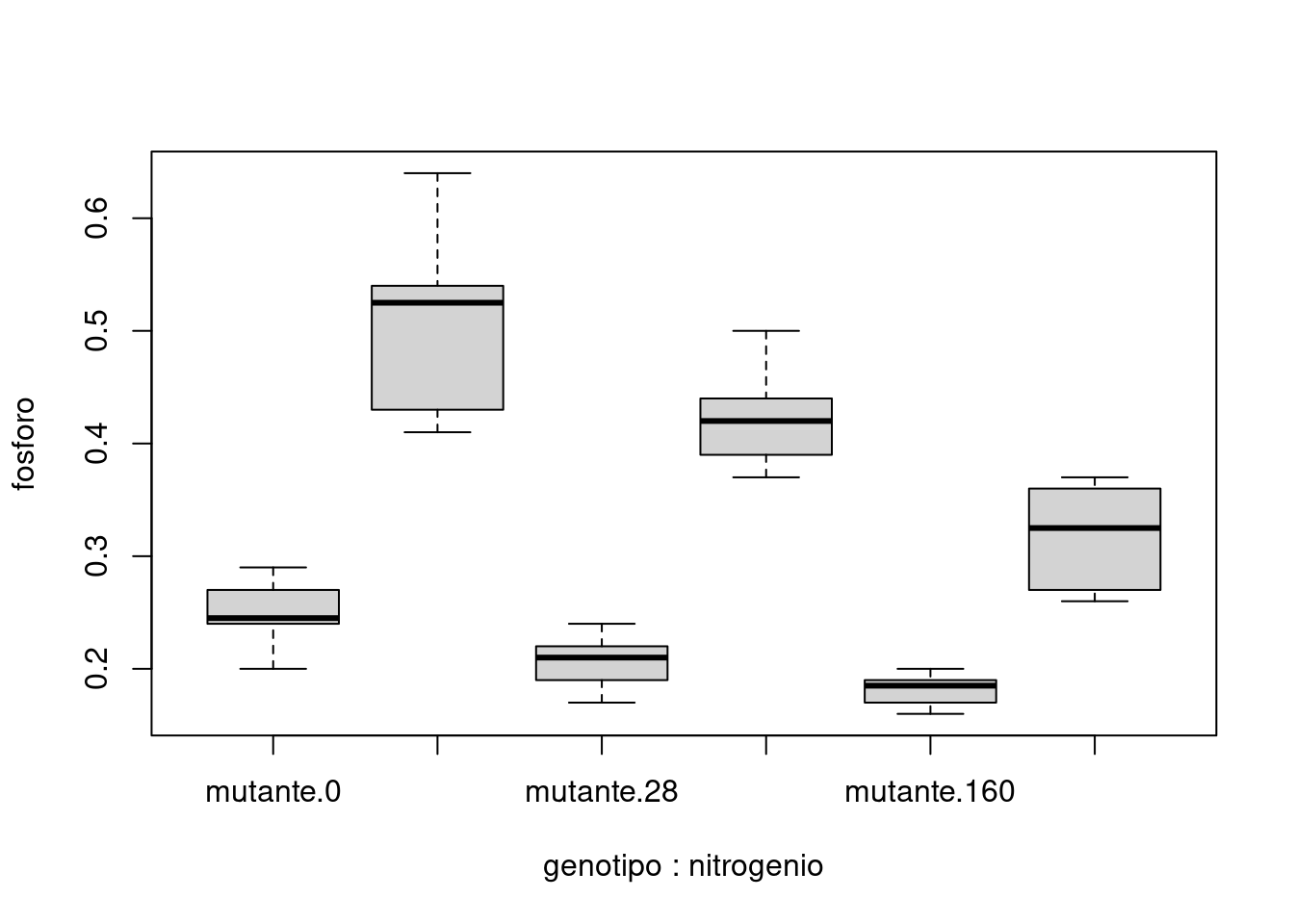
## Gráfico da interação
with(tomate, interaction.plot(genotipo, nitrogenio, fosforo))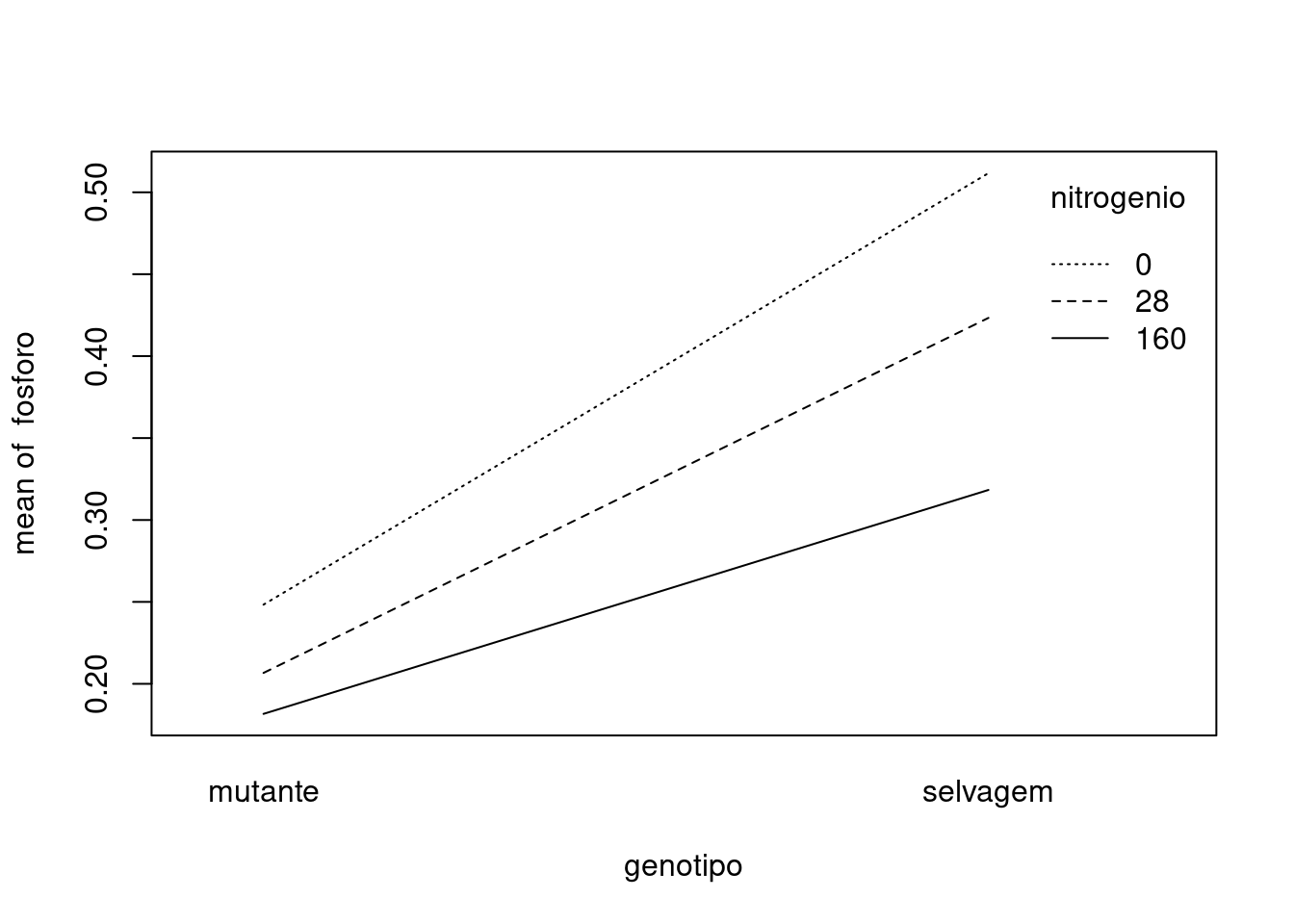
with(tomate, interaction.plot(nitrogenio, genotipo, fosforo))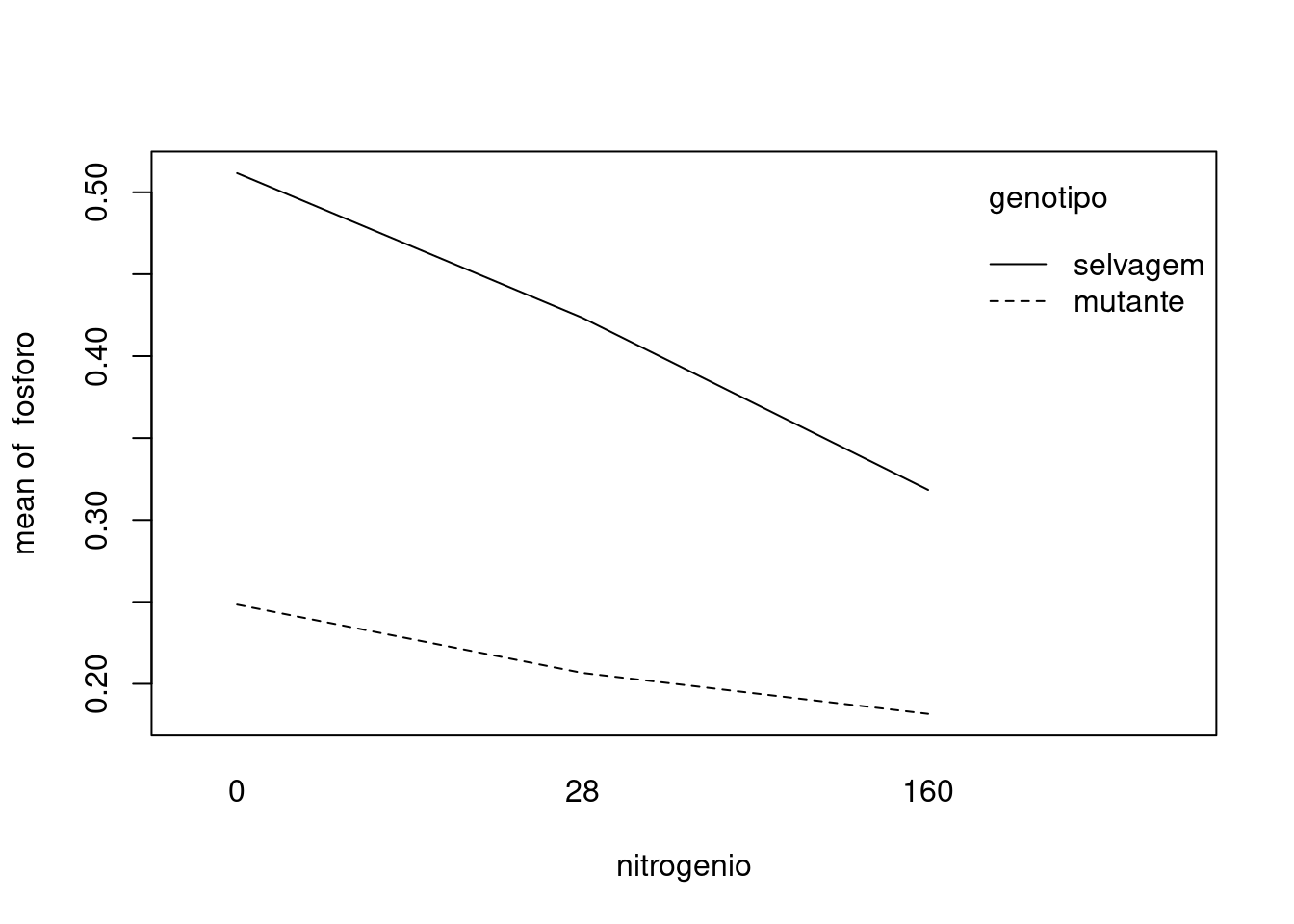
Efetuamos a Análise de Variância para os efeitos principais e para a interação:
## Analysis of Variance Table
##
## Response: fosforo
## Df Sum Sq Mean Sq F value Pr(>F)
## genotipo 1 0.38028 0.38028 177.148 4.019e-14 ***
## nitrogenio 2 0.10140 0.05070 23.618 6.911e-07 ***
## genotipo:nitrogenio 2 0.02462 0.01231 5.735 0.007777 **
## Residuals 30 0.06440 0.00215
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1A interação é significativa (p-valor = 0,0077), mas ela é pequena comparada com os efeitos principais.
O efeito principal para genótipos (p-valor < 0,001) nos mostra que tomates selvagens com micorrizas tem maiores níveis de fósforo para todas as doses de nitrogênio, devido ao benefício da simbiose.
O efeito principal para nitrogênio (p-valor < 0,001) mostra que os níveis de fósforo diminuem com o aumento da dose de nitrogênio.
Verificação dos presupostos da Normalidade e Homogeneidade das Variâncias (análise não mostrada): não há evidência de violação dos pressupostos.
Teste de comparação de médias para o efeito principal do fator nitrogênio:
tk_nitrogenio <- emmeans::emmeans(aov_tomate, ~nitrogenio, contr = "tukey")## NOTE: Results may be misleading due to involvement in interactions
## contrastes
tk_nitrogenio$contrasts## contrast estimate SE df t.ratio p.value
## nitrogenio0 - nitrogenio28 0.065 0.0189 30 3.436 0.0048
## nitrogenio0 - nitrogenio160 0.130 0.0189 30 6.873 <.0001
## nitrogenio28 - nitrogenio160 0.065 0.0189 30 3.436 0.0048
##
## Results are averaged over the levels of: genotipo
## P value adjustment: tukey method for comparing a family of 3 estimates## contrast estimate SE df lower.CL upper.CL
## nitrogenio0 - nitrogenio28 0.065 0.0189 30 0.0184 0.112
## nitrogenio0 - nitrogenio160 0.130 0.0189 30 0.0834 0.177
## nitrogenio28 - nitrogenio160 0.065 0.0189 30 0.0184 0.112
##
## Results are averaged over the levels of: genotipo
## Confidence level used: 0.95
## Conf-level adjustment: tukey method for comparing a family of 3 estimates## nitrogenio emmean SE df lower.CL upper.CL .group
## 160 0.250 0.0134 30 0.223 0.277 a
## 28 0.315 0.0134 30 0.288 0.342 b
## 0 0.380 0.0134 30 0.353 0.407 c
##
## Results are averaged over the levels of: genotipo
## Confidence level used: 0.95
## P value adjustment: tukey method for comparing a family of 3 estimates
## significance level used: alpha = 0.05
## NOTE: If two or more means share the same grouping symbol,
## then we cannot show them to be different.
## But we also did not show them to be the same.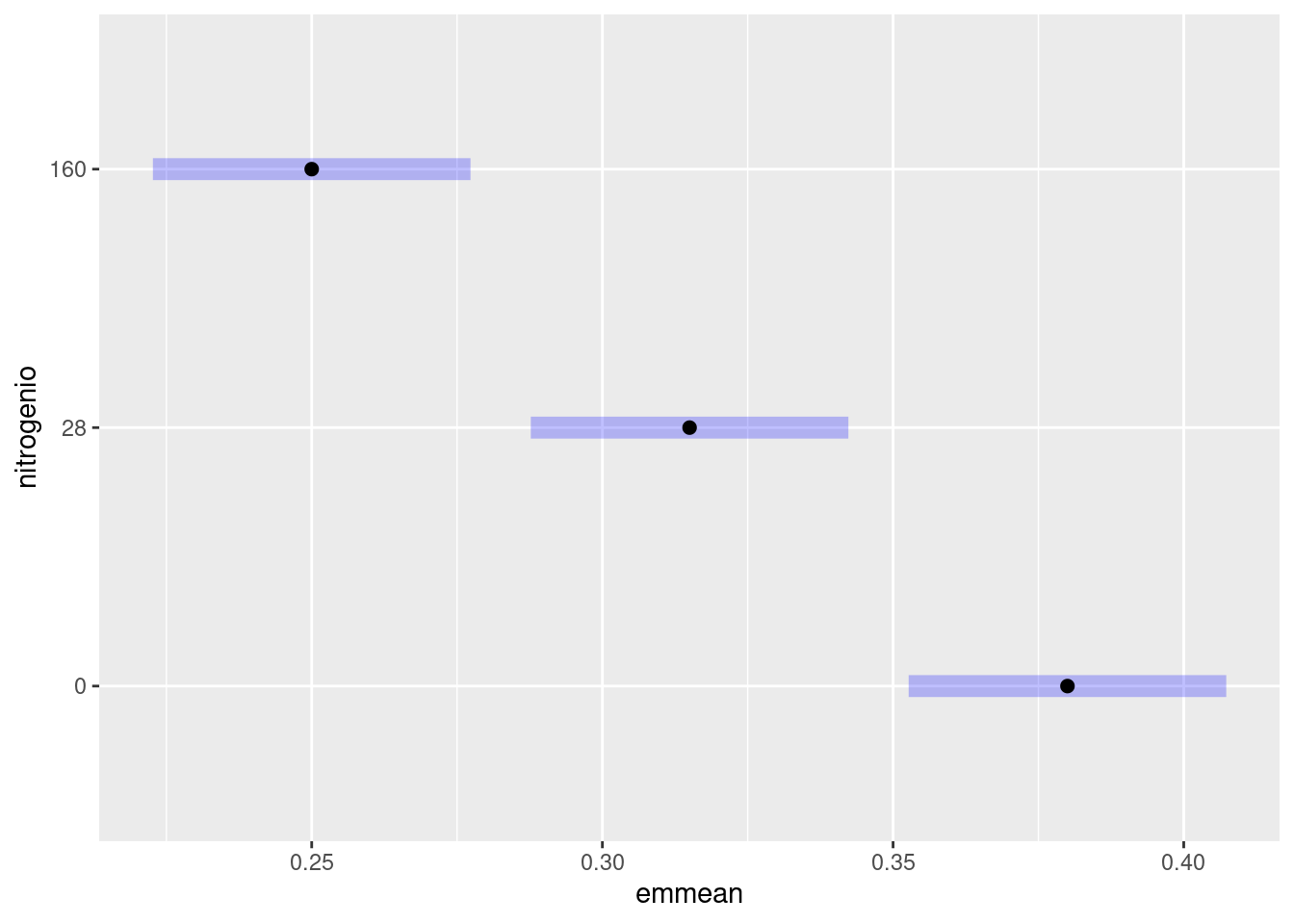
Teste de comparação de médias para o efeito principal do fator genótipo:
tk_genotipo <- emmeans::emmeans(aov_tomate, ~genotipo, contr = "tukey")## NOTE: Results may be misleading due to involvement in interactions
## contrastes
tk_genotipo$contrasts## contrast estimate SE df t.ratio p.value
## mutante - selvagem -0.206 0.0154 30 -13.310 <.0001
##
## Results are averaged over the levels of: nitrogenio## contrast estimate SE df lower.CL upper.CL
## mutante - selvagem -0.206 0.0154 30 -0.237 -0.174
##
## Results are averaged over the levels of: nitrogenio
## Confidence level used: 0.95## genotipo emmean SE df lower.CL upper.CL .group
## mutante 0.212 0.0109 30 0.190 0.235 a
## selvagem 0.418 0.0109 30 0.395 0.440 b
##
## Results are averaged over the levels of: nitrogenio
## Confidence level used: 0.95
## significance level used: alpha = 0.05
## NOTE: If two or more means share the same grouping symbol,
## then we cannot show them to be different.
## But we also did not show them to be the same.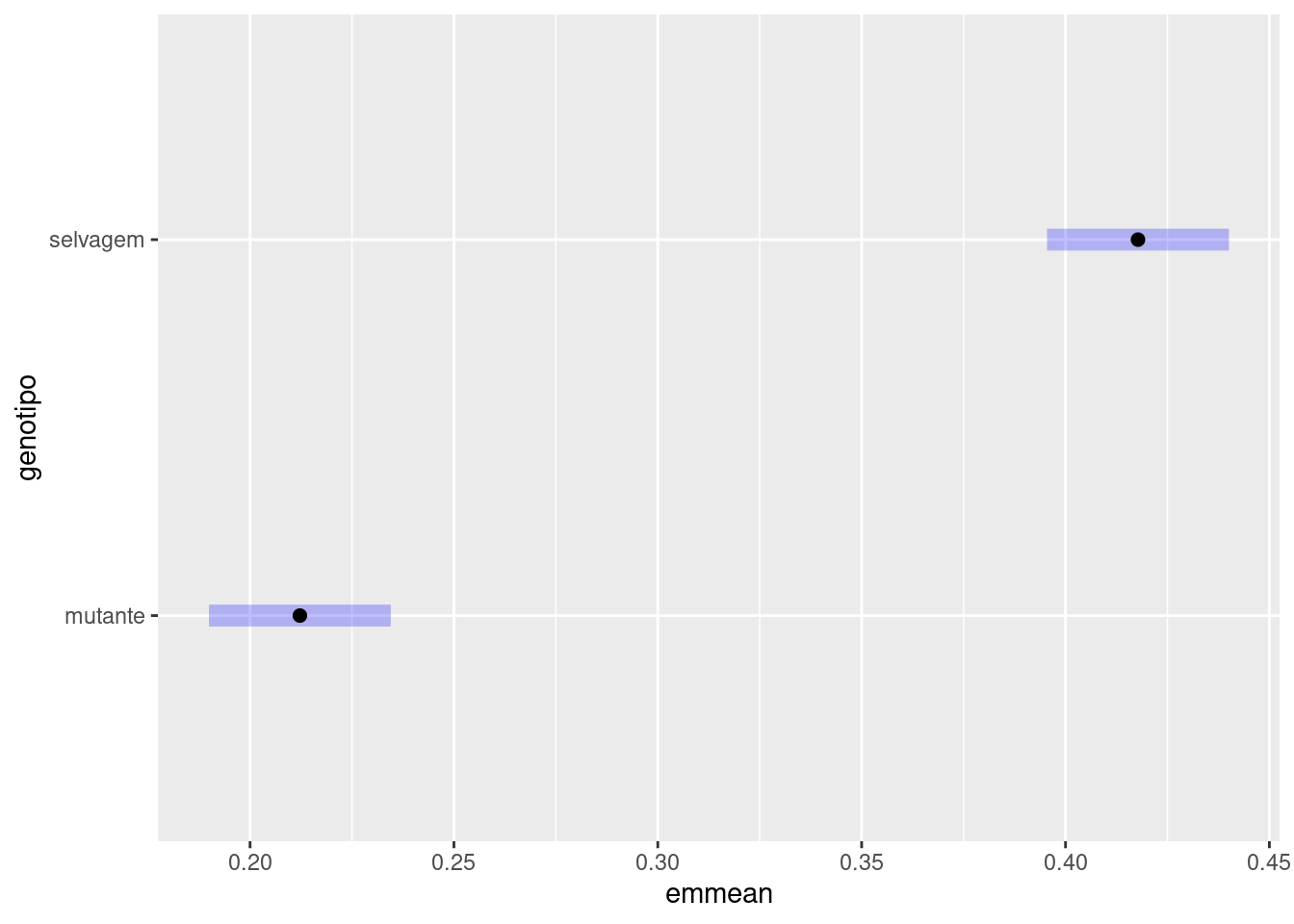
Conclusão:
Tomates selvagens com micorrizas tem níveis maiores de fósforo que mutantes sem micorrizas.
O nitrogênio reduz o nível de fosforo nas plantas.
A redução é maior nas plantas selvagens, mas esta interação não é muito grande em termos práticos.
Por último, temos um exemplo em que há uma forte interação que faz os efeitos principais sem sentido. Uma Anova de dois fatores com um efeito de interação forte é frequentemente mais difícil de interpretar.
Exemplo 11.3 (Análise de Variância para dois fatores - interação significativa) Milho com maior conteúdo de aminoácidos pode ser vantajoso na alimentação animal. Nove tratamentos foram arranjados em um fatorial 3x3:
- Milho normal e duas variedades alteradas: opaque-2 e floury-2
- Doses de proteína na dieta: 20, 16 e 12%
As dietas milho+proteína foram usadas na alimentação de 90 pintinhos machos de 1 dia. A variável resposta é o peso (em gramas) após 21 dias milho.xlsx.
Qual combinação de milho e proteína leva ao maior aumento de peso?
Há três grupos de hipóteses serem testadas:
H0 : não há interação entre as variedade de milho e a quantidade de proteína.
H1: há interação entre as variedade de milho e a quantidade de proteína.
H0: a quantidade de proteína não influencia no peso dos pintinhos.
H1: a quantidade de proteína influencia no peso dos pintinhos.
H0: a variedade de milho não influencia no peso dos pintinhos.
H1: a variedade de milho influencia no peso dos pintinhos.
Análise exploratória:
milho <- readxl::read_excel("data/milho.xlsx") %>%
mutate(
variedade = factor(variedade),
proteina = factor(proteina)
)
milho %>%
group_by(variedade, proteina) %>%
summarise(
n = n(),
media = mean(peso),
desvpad = sd(peso),
var = var(peso)
)## `summarise()` has grouped output by 'variedade'. You can override using the
## `.groups` argument.## # A tibble: 9 × 6
## # Groups: variedade [3]
## variedade proteina n media desvpad var
## <fct> <fct> <int> <dbl> <dbl> <dbl>
## 1 flou 12 10 271. 32.1 1033.
## 2 flou 16 10 434. 49.7 2474.
## 3 flou 20 10 465. 49.2 2416.
## 4 norm 12 10 195. 50.5 2553.
## 5 norm 16 10 389. 48.1 2314.
## 6 norm 20 10 486. 38.7 1496.
## 7 opaq 12 10 264. 26.0 675.
## 8 opaq 16 10 339. 54.4 2959.
## 9 opaq 20 10 438. 61.2 3741.
## Boxplot - Efeito principal fator A
boxplot(peso ~ variedade, data = milho)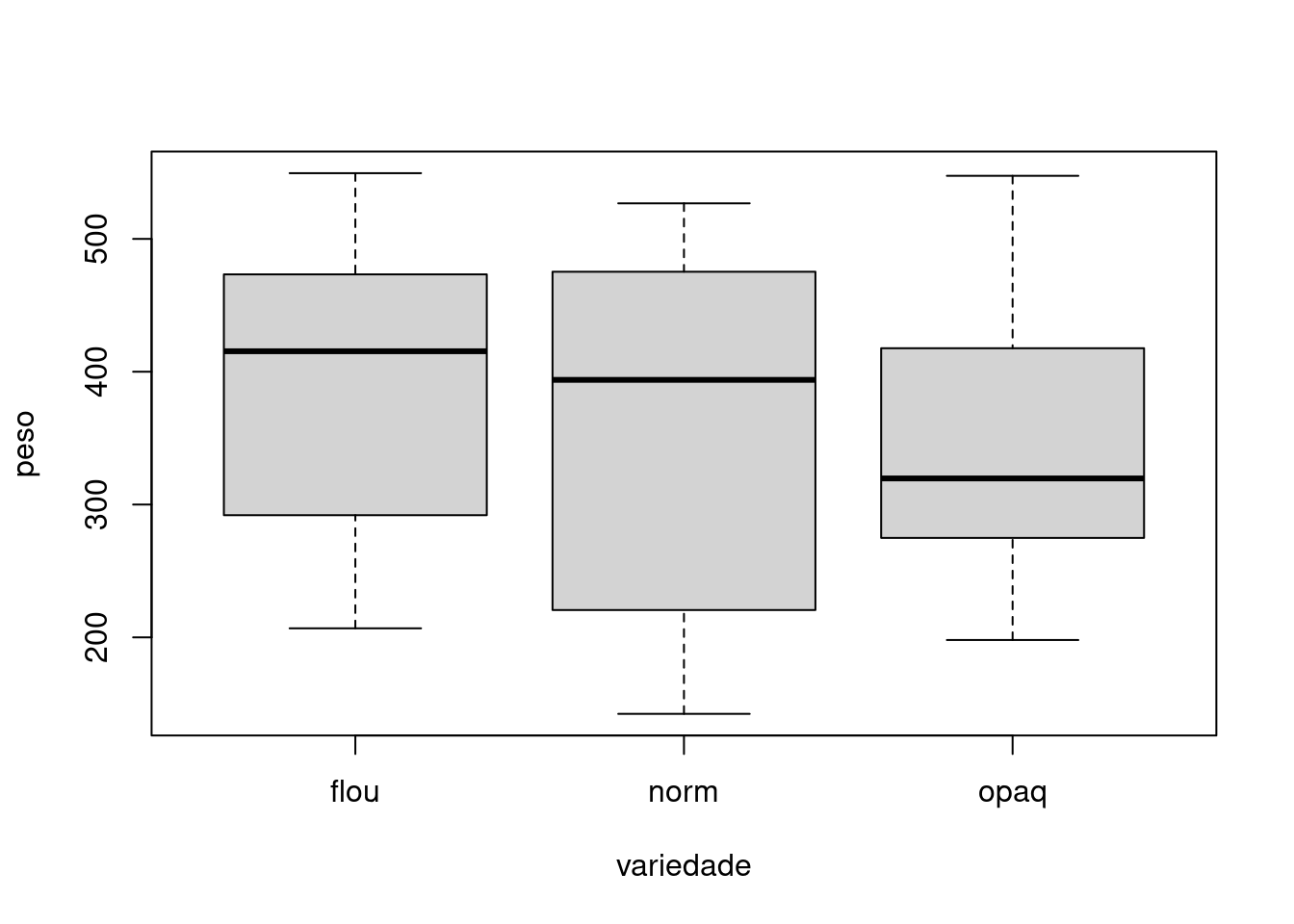
## Boxplot - Efeito principal fator B
boxplot(peso ~ proteina, data = milho)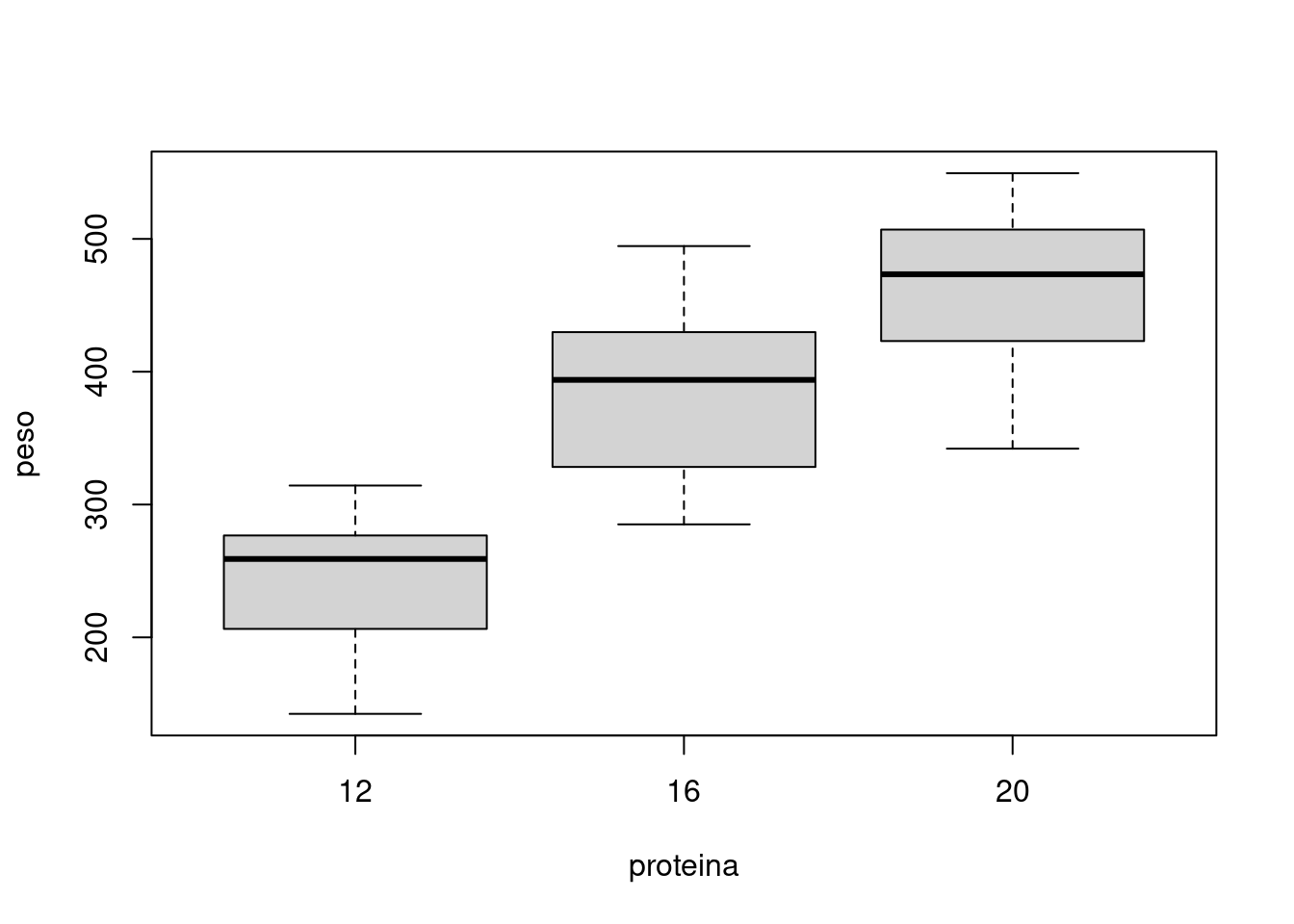
## Boxplot - Interação
boxplot(peso ~ variedade * proteina, data = milho)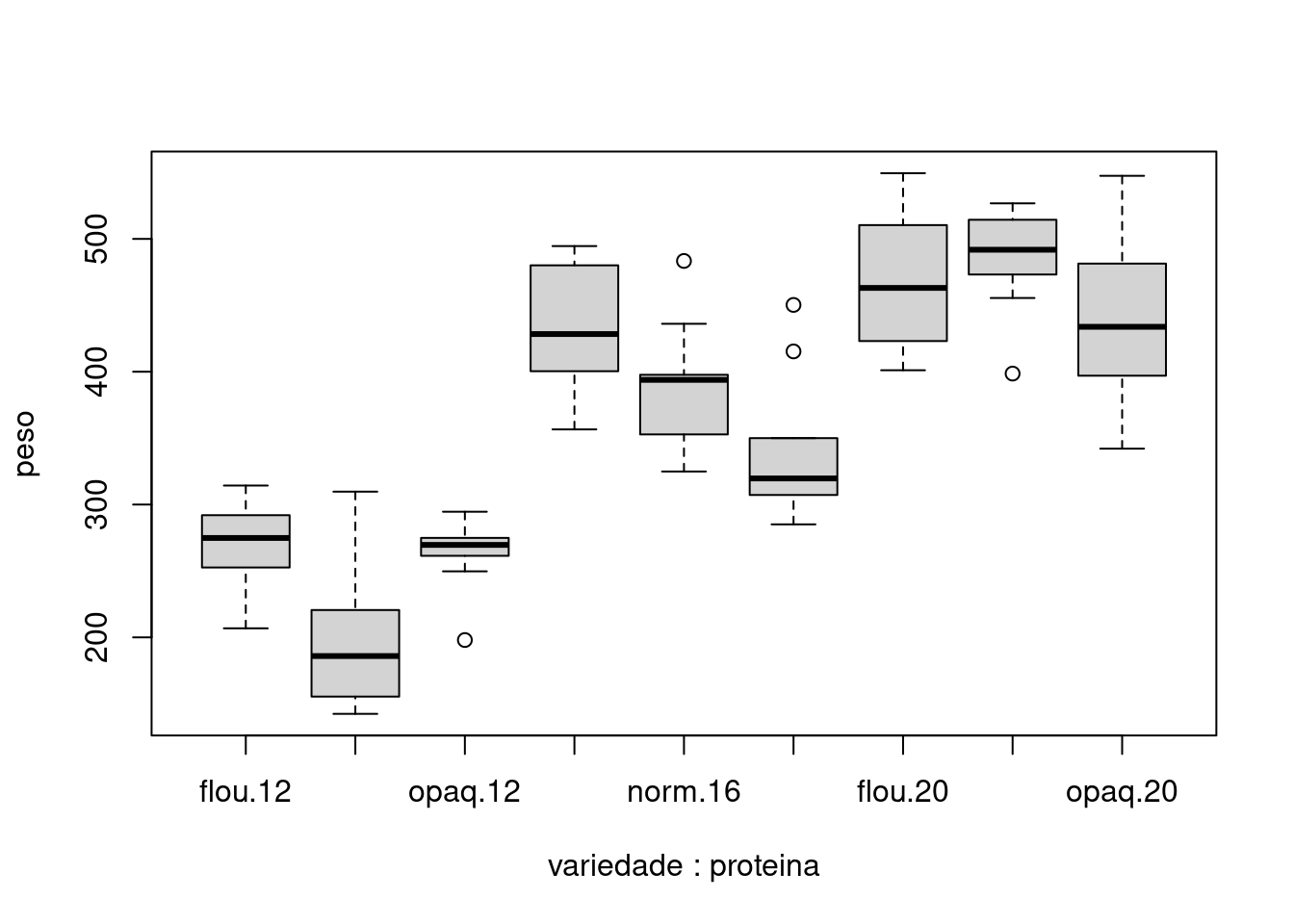
## Gráfico da interação
with(milho, interaction.plot(variedade, proteina, peso))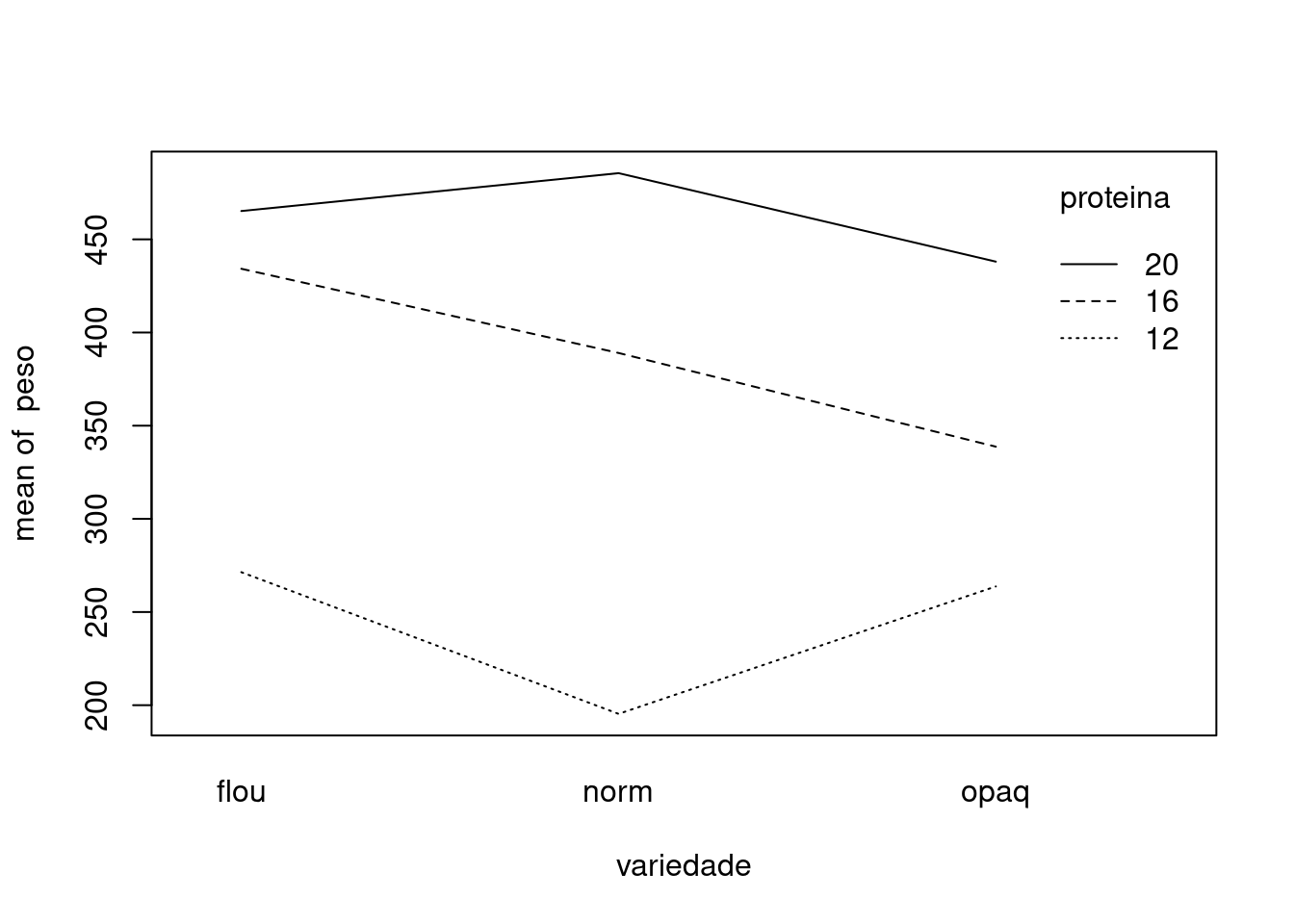
with(milho, interaction.plot(proteina, variedade, peso))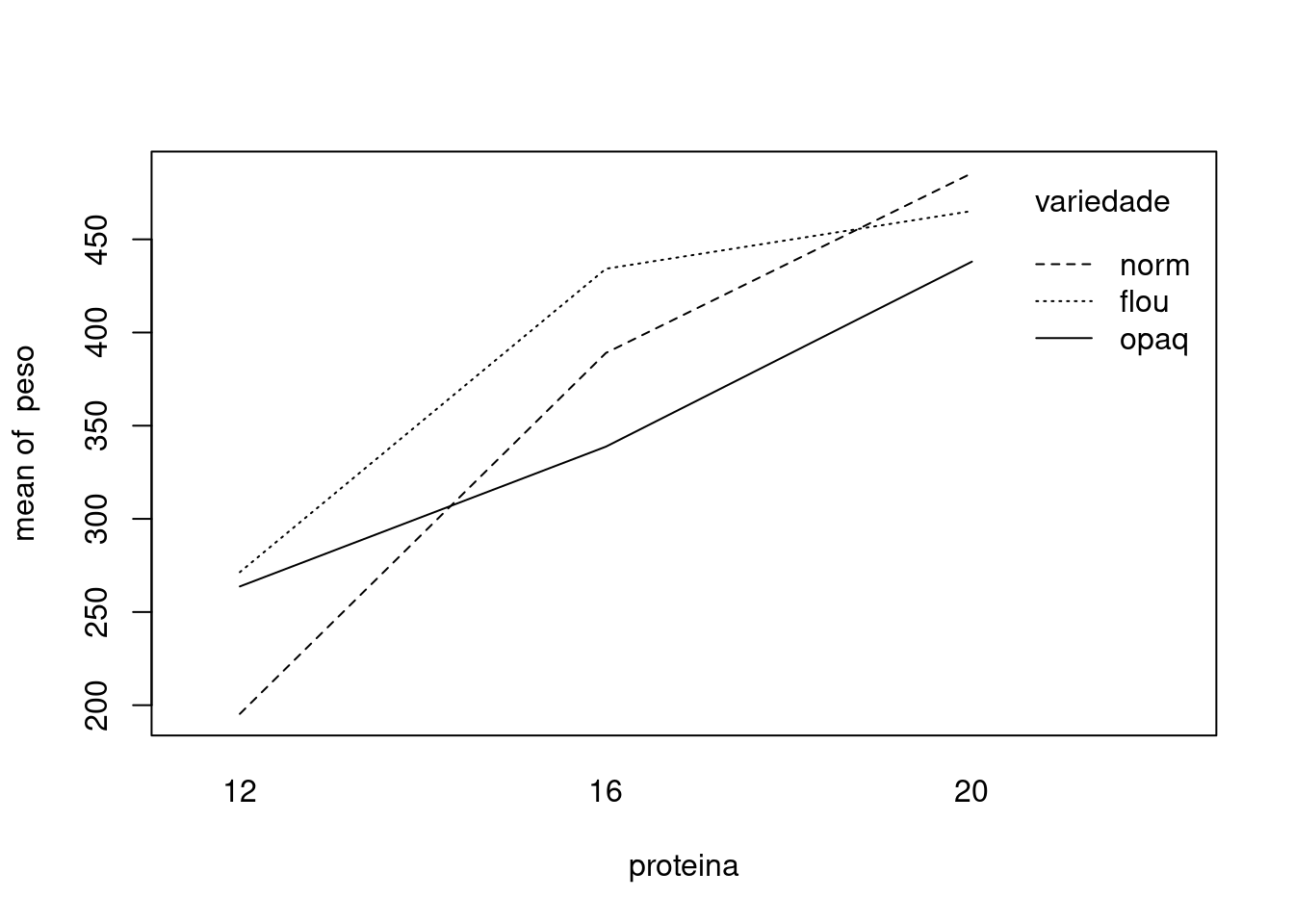
Efetuamos a Análise de Variância para os efeitos principais e para a interação:
## Analysis of Variance Table
##
## Response: peso
## Df Sum Sq Mean Sq F value Pr(>F)
## variedade 2 31137 15568 7.1265 0.00141 **
## proteina 2 745621 372810 170.6557 < 2.2e-16 ***
## variedade:proteina 4 60894 15223 6.9686 7.143e-05 ***
## Residuals 81 176951 2185
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Verificação dos presupostos da Normalidade e Homogeneidade das Variâncias (análise não mostrada): não há evidência de violação dos pressupostos.
Desdobramento da interação: variedade dentro de cada nível de proteína.
## Desdobramento da interação
phia::testInteractions(aov_milho, fixed="proteina", across="variedade")## F Test:
## P-value adjustment method: holm
## variedade1 variedade2 Df Sum of Sq F Pr(>F)
## 12 7.66 -68.39 2 35065 8.0256 0.0013218 **
## 16 95.47 50.28 2 45616 10.4404 0.0002773 ***
## 20 27.16 47.48 2 11350 2.5977 0.0806380 .
## Residuals 81 176951
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## Teste de Tukey para a interação
tk_variedade <- emmeans::emmeans(aov_milho, ~proteina|variedade, contr = "tukey")
## Contrastes
tk_variedade$contrasts## variedade = flou:
## contrast estimate SE df t.ratio p.value
## proteina12 - proteina16 -162.8 20.9 81 -7.790 <.0001
## proteina12 - proteina20 -193.8 20.9 81 -9.273 <.0001
## proteina16 - proteina20 -31.0 20.9 81 -1.483 0.3046
##
## variedade = norm:
## contrast estimate SE df t.ratio p.value
## proteina12 - proteina16 -193.7 20.9 81 -9.266 <.0001
## proteina12 - proteina20 -290.2 20.9 81 -13.883 <.0001
## proteina16 - proteina20 -96.5 20.9 81 -4.617 <.0001
##
## variedade = opaq:
## contrast estimate SE df t.ratio p.value
## proteina12 - proteina16 -75.0 20.9 81 -3.589 0.0016
## proteina12 - proteina20 -174.3 20.9 81 -8.340 <.0001
## proteina16 - proteina20 -99.3 20.9 81 -4.751 <.0001
##
## P value adjustment: tukey method for comparing a family of 3 estimates## variedade = flou:
## contrast estimate SE df lower.CL upper.CL
## proteina12 - proteina16 -162.8 20.9 81 -212.7 -112.9
## proteina12 - proteina20 -193.8 20.9 81 -243.7 -143.9
## proteina16 - proteina20 -31.0 20.9 81 -80.9 18.9
##
## variedade = norm:
## contrast estimate SE df lower.CL upper.CL
## proteina12 - proteina16 -193.7 20.9 81 -243.6 -143.8
## proteina12 - proteina20 -290.2 20.9 81 -340.1 -240.3
## proteina16 - proteina20 -96.5 20.9 81 -146.4 -46.6
##
## variedade = opaq:
## contrast estimate SE df lower.CL upper.CL
## proteina12 - proteina16 -75.0 20.9 81 -124.9 -25.1
## proteina12 - proteina20 -174.3 20.9 81 -224.2 -124.4
## proteina16 - proteina20 -99.3 20.9 81 -149.2 -49.4
##
## Confidence level used: 0.95
## Conf-level adjustment: tukey method for comparing a family of 3 estimates## variedade = flou:
## proteina emmean SE df lower.CL upper.CL .group
## 12 271 14.8 81 242 301 a
## 16 434 14.8 81 405 464 b
## 20 465 14.8 81 436 495 b
##
## variedade = norm:
## proteina emmean SE df lower.CL upper.CL .group
## 12 195 14.8 81 166 225 a
## 16 389 14.8 81 360 418 b
## 20 486 14.8 81 456 515 c
##
## variedade = opaq:
## proteina emmean SE df lower.CL upper.CL .group
## 12 264 14.8 81 234 293 a
## 16 339 14.8 81 309 368 b
## 20 438 14.8 81 409 467 c
##
## Confidence level used: 0.95
## P value adjustment: tukey method for comparing a family of 3 estimates
## significance level used: alpha = 0.05
## NOTE: If two or more means share the same grouping symbol,
## then we cannot show them to be different.
## But we also did not show them to be the same.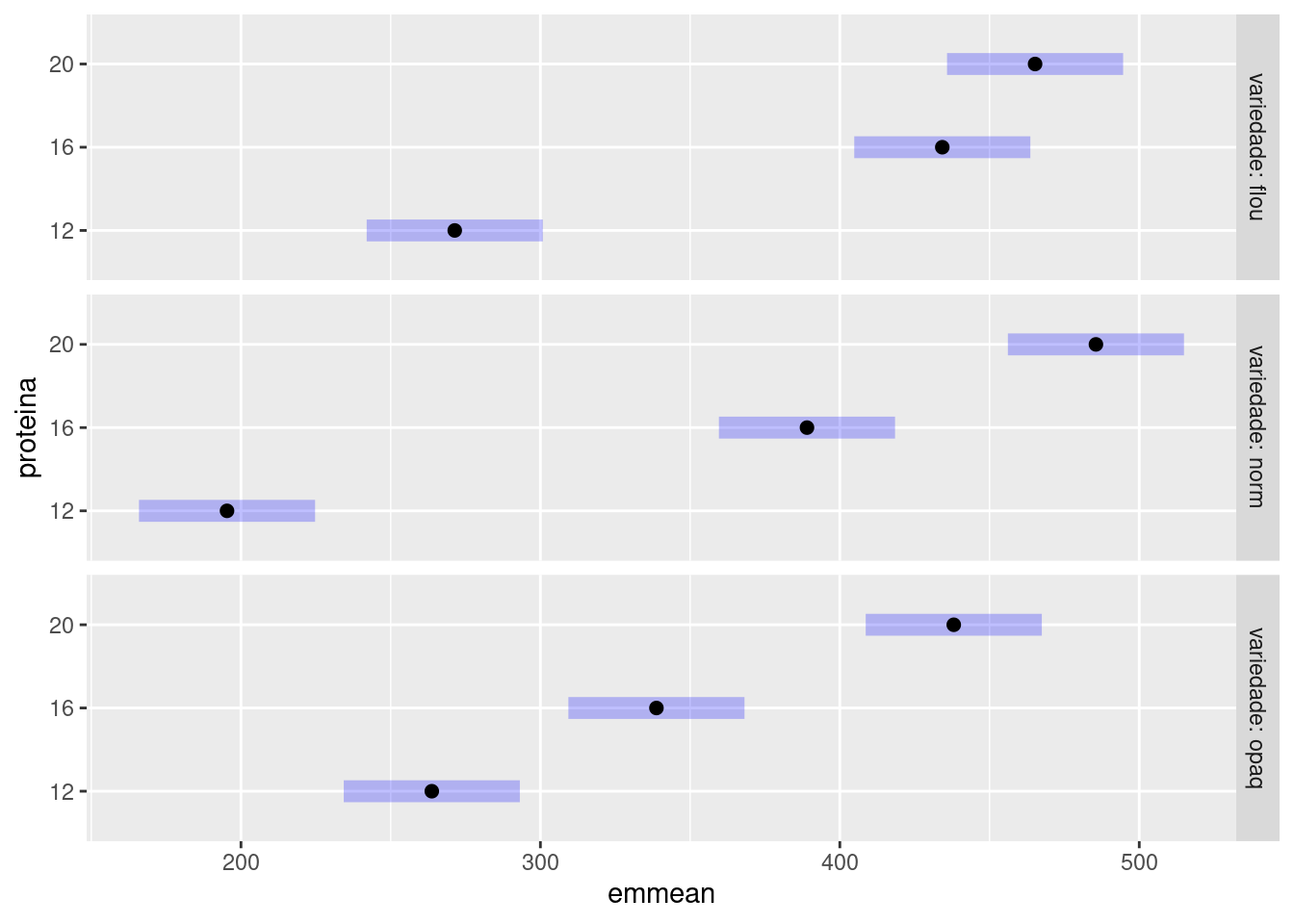
Desdobramento da interação: proteína dentro de cada nível de variedade.
## Desdobramento da interação
phia::testInteractions(aov_milho, fixed="variedade", across="proteina")## F Test:
## P-value adjustment method: holm
## proteina1 proteina2 Df Sum of Sq F Pr(>F)
## flou -193.82 -30.99 2 216801 49.621 1.708e-14 ***
## norm -290.19 -96.50 2 436794 99.972 < 2.2e-16 ***
## opaq -174.32 -99.30 2 152920 35.000 1.109e-11 ***
## Residuals 81 176951
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## Teste de Tukey para a interação
tk_proteina <- emmeans::emmeans(aov_milho, ~variedade|proteina, contr = "tukey")
## Contrastes
tk_proteina$contrasts## proteina = 12:
## contrast estimate SE df t.ratio p.value
## flou - norm 76.05 20.9 81 3.638 0.0014
## flou - opaq 7.66 20.9 81 0.366 0.9287
## norm - opaq -68.39 20.9 81 -3.272 0.0044
##
## proteina = 16:
## contrast estimate SE df t.ratio p.value
## flou - norm 45.19 20.9 81 2.162 0.0840
## flou - opaq 95.47 20.9 81 4.567 0.0001
## norm - opaq 50.28 20.9 81 2.405 0.0479
##
## proteina = 20:
## contrast estimate SE df t.ratio p.value
## flou - norm -20.32 20.9 81 -0.972 0.5965
## flou - opaq 27.16 20.9 81 1.299 0.3997
## norm - opaq 47.48 20.9 81 2.271 0.0657
##
## P value adjustment: tukey method for comparing a family of 3 estimates## proteina = 12:
## contrast estimate SE df lower.CL upper.CL
## flou - norm 76.05 20.9 81 26.144 126.0
## flou - opaq 7.66 20.9 81 -42.246 57.6
## norm - opaq -68.39 20.9 81 -118.296 -18.5
##
## proteina = 16:
## contrast estimate SE df lower.CL upper.CL
## flou - norm 45.19 20.9 81 -4.716 95.1
## flou - opaq 95.47 20.9 81 45.564 145.4
## norm - opaq 50.28 20.9 81 0.374 100.2
##
## proteina = 20:
## contrast estimate SE df lower.CL upper.CL
## flou - norm -20.32 20.9 81 -70.226 29.6
## flou - opaq 27.16 20.9 81 -22.746 77.1
## norm - opaq 47.48 20.9 81 -2.426 97.4
##
## Confidence level used: 0.95
## Conf-level adjustment: tukey method for comparing a family of 3 estimates## proteina = 12:
## variedade emmean SE df lower.CL upper.CL .group
## norm 195 14.8 81 166 225 a
## opaq 264 14.8 81 234 293 b
## flou 271 14.8 81 242 301 b
##
## proteina = 16:
## variedade emmean SE df lower.CL upper.CL .group
## opaq 339 14.8 81 309 368 a
## norm 389 14.8 81 360 418 b
## flou 434 14.8 81 405 464 b
##
## proteina = 20:
## variedade emmean SE df lower.CL upper.CL .group
## opaq 438 14.8 81 409 467 a
## flou 465 14.8 81 436 495 a
## norm 486 14.8 81 456 515 a
##
## Confidence level used: 0.95
## P value adjustment: tukey method for comparing a family of 3 estimates
## significance level used: alpha = 0.05
## NOTE: If two or more means share the same grouping symbol,
## then we cannot show them to be different.
## But we also did not show them to be the same.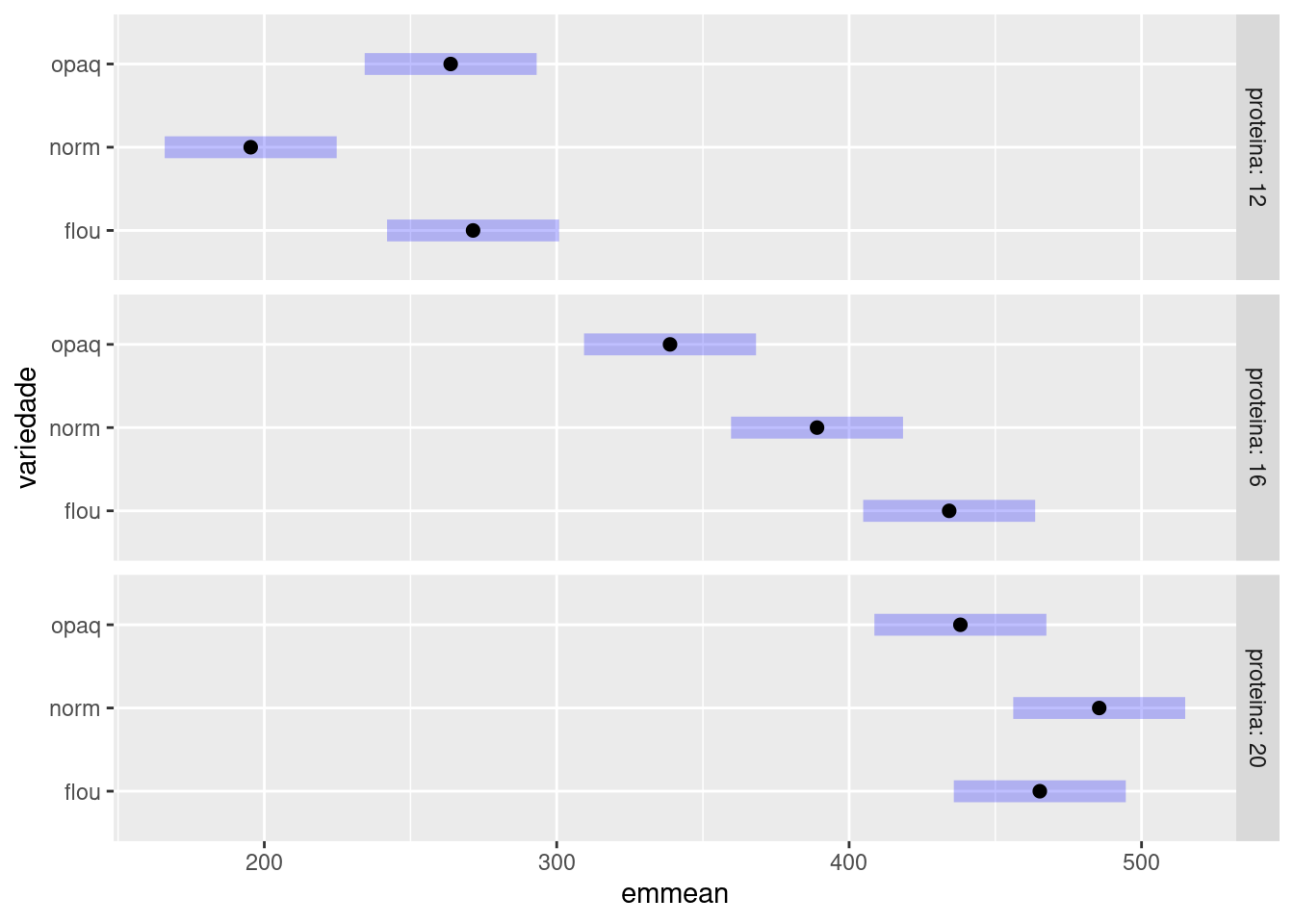
Há um efeito importante da interação:
O milho normal é o pior para 12% de proteína e o melhor para 20%. Floury é o melhor para 12% e 16% de proteína. Opaque é sempre inferior ao Floury, mas melhor que o normal somente para 12% de proteína.
Apesar de haver um efeito principal para variedade e proteína, estes são menos importantes devido a interação.
Conclusão:
- Maior dose de proteína ajuda a acelerar o ganho de peso.
- Os três tipos de milho não diferem quando a dieta tem 20% de proteína.
- Floury é melhor quando a dose de proteína é de 16% e 20%.