
Capítulo 2 Começando com o R
Neste capítulo, vamos iniciar com o uso do R e do RStudio.
2.1 A interface do RStudio
O RStudio possui uma interface simples mas bastante funcional para trabalhar com códigos do R.
São quatro janelas principais como pode ser visto na Figura 2.1.
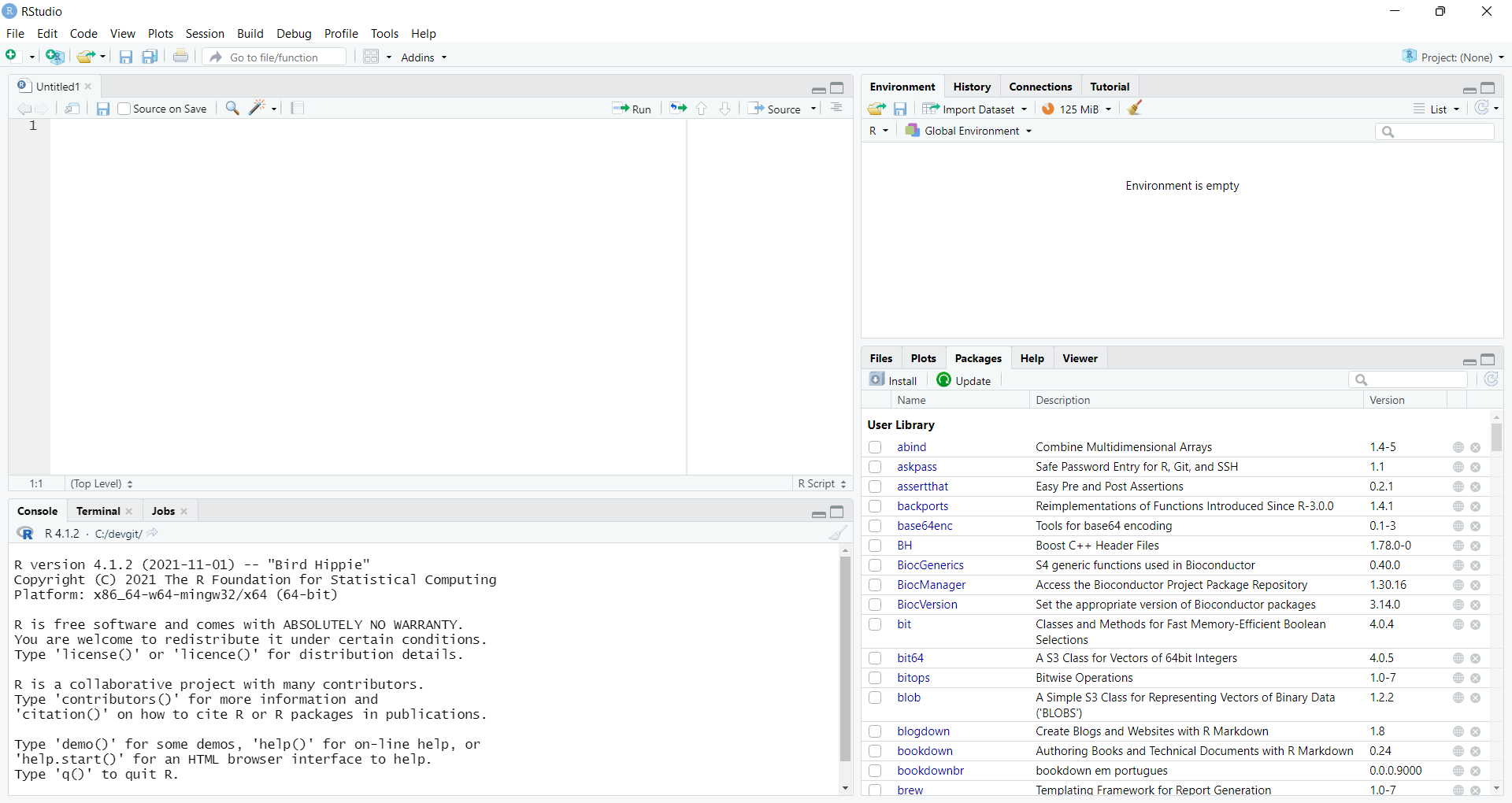
Figura 2.1: Interface do RStudio
Source (Editor) (canto superior esquerdo): local para editar scripts, que são sequências de comandos que podem ser salvos e utilizados repetidamente. Os comandos desta janela são executados clicando no botão Run ou pelo atalho do teclado +. Caso o editor não esteja aparecendo, clique em File -> New File -> R Script, ou pelo atalho ++
Console (canto inferior esquerdo): local para digitar e executar os comandos, assim como obter o resultado destes. Também mostra o resutado dos comando executados a partir do editor.
Environment (Ambiente) (canto superior direito): local que mostra as variáveis definidas, entre outros.
Files/Plots/Packages/Help/Viewer (canto inferior direito): local multiuso com várias abas, que mostra e configura várias opções, como arquivos, gráficos, pacotes, ajuda e visualização.
2.2 Criar um projeto no RStudio
Um projeto do RStudio mantem todos os arquivos relevantes para a análise, como banco de dados, scripts, resultados e figuras. É uma excelente maneira de evitar ter de lidar com caminhos e diretórios.
Para começar um novo projeto, clique em File -> New Project e siga os passos nas janelas conforme aparecerem:
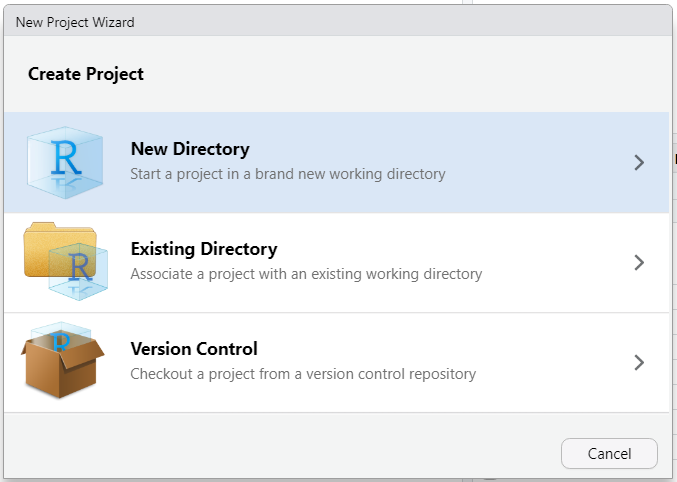
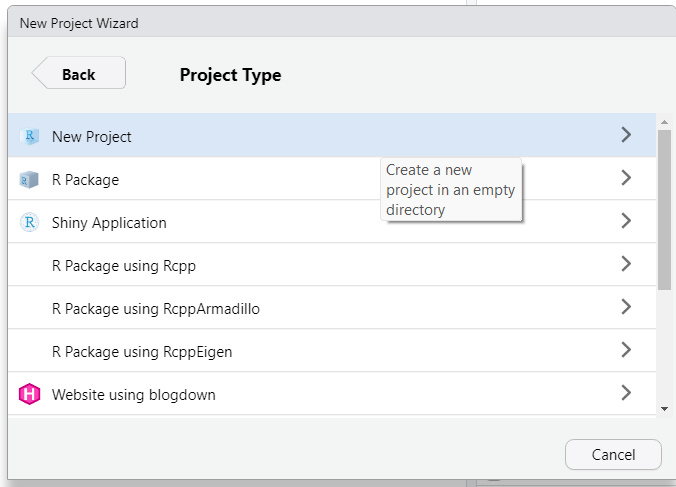
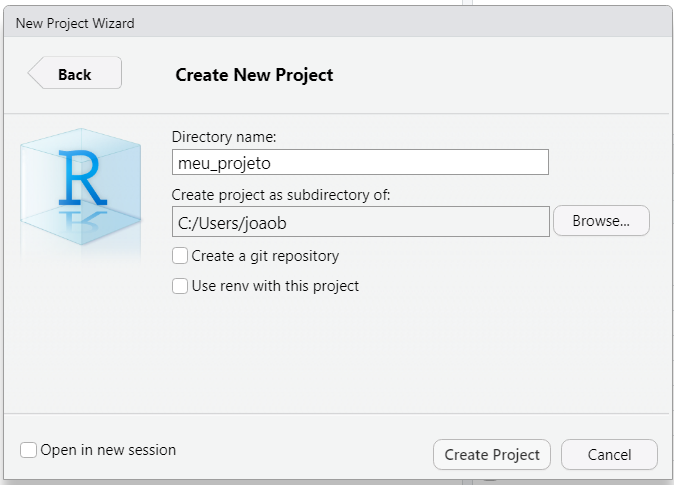
Dê um bom nome para seu novo projeto e cuide do local em que este será salvo.
2.3 Primeiros códigos
Após o projeto criado, vamos digitar os primeiros códigos na janela do Script. Tente digitar o seguinte:
1 + 1Em seguida, posicione o cursor na linha que acabou de digitar, aperte o botao Run ou + e veja o resultado.
1 + 1## [1] 2Esta foi uma operação muito simples, então vamos ver alguns códigos mais interessantes.
Para criar um objeto e atribuir um valor a ele, primeiro precisamos dar um nome9 ao objeto, seguido do símbolo de atribuição <- (atalho: + ) 10 e do valor que desejamos atribuir ao objeto:
x <- 10 # leia-se: x recebe o valor 10Neste exemplo, o objeto denominado xrecebeu o valor 10. Notem que ao executar este comando, o objeto recém criado aparece na janela Environment, confirmando a criação do objeto.
Este exemplo também mostrou como usar um comentário, que é um texto não interpretado pelo programa. Os comentários são bastante úteis para explicar as linhas de código. Os comentários são escritos após o símbolo #. Para comentar uma ou várias linhas, selecione o texto e aperte ++.
Para verificar o valor que está atribuído a um objeto, basta digitar o nome do objeto11:
x## [1] 10Um objeto pode ter seu valor modificado ou copiado com uma nova atribuição:
y <- x
x <- x * 2
z <- x + y
y## [1] 10
x## [1] 20
z## [1] 30Observe que a ordem de execução dos comandos é importante no resultado.
Um dos objetos mais útil na programação em R é o vetor, que pode ser entendido como uma série de valores armazenada em um objeto. Para criar um vetor, utilizamos a função c():
primos6 <- c(2, 3, 5, 7, 11, 13)
primos6## [1] 2 3 5 7 11 13A função str nos mostra a estrutura do vetor:
str(primos6)## num [1:6] 2 3 5 7 11 13indicando que o objeto primos6 é um vetor numérico (num).
Uma parte do vetor pode ser extraída como uso de colchetes [ ]:
primos6[3] # acessa o terceiro elemento do vetor## [1] 5
primos6[4:6] # acessa do 4° ao 6° elemento do vetor## [1] 7 11 13As operações com vetores são aplicadas a todos os elementos dele:
primos6^2## [1] 4 9 25 49 121 169Calcular a média dos valores deste vetor com a função mean:
mean(primos6)## [1] 6.833333Muitas funções são disponibilizadas em pacotes. Para acessar uma função de um pacote usamos a sintaxe pacote::função. Por exemplo, para acessar as funções first e last do pacote dplyr:
dplyr::first(primos6)## [1] 2
dplyr::last(primos6)## [1] 13Alternativamente, é possível carregar o pacote com a função library e depois usar as funções diretamente:
##
## Attaching package: 'dplyr'## The following objects are masked from 'package:stats':
##
## filter, lag## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, union
first(primos6)## [1] 2
last(primos6)## [1] 13Para criar uma gráfico, podemos usar várias das funções base do R para esse fim. Neste exemplo, a função hist cria um gráfico do tipo histograma para um conjunto de 10.000 valores gerados aleatoriamente pela função rnorm.
Na seção 5 veremos como utilizar o pacote ggplot2 para criar gráficos mais elaborados.
2.4 Criar uma função no R
Funções são usadas para dividir o código em partes mais simples, mas fáceis de manter e de entender. As funções também são facilmente reutilizadas.
Para criar uma função no R, a sintaxe é a seguinte:
nome_funcao <- function (argumentos) {
declaração
}Como exemplo, vamos criar uma função que simula o lançamento dado: dado6
dado6 <- function(n) {
sample(1:6, size = n, replace = TRUE)
}Esta função retorna n valores entre 1 e 6 com substituição.
Para utilizar a função recém criada, chamamos ela pelo nome e passamos um valor para os argumentos:
dado6(n = 1)## [1] 1
dado6(n = 3)## [1] 4 3 22.5 Data-frame
O data frame é uma estrutura bidimensional em que cada componente forma uma coluna e o conteúdo de cada componente forma as linhas. Em um data frame, é importante que todas as colunas tenham o mesmo comprimento, de modo que a estrutura de dados resulte em um formato retangular. Também podemos imaginá-lo como um agrupamento de vetores de mesmo tamanho.
Para criá-lo, usamos a função data.frame
df <- data.frame(
x = c("a", "b", "c"),
y = c(16, 32, 64)
)
df## x y
## 1 a 16
## 2 b 32
## 3 c 64Idem ao anterior, mas com os vetores sendo criado anteriormente e separadamente
x <- c("a", "b", "c")
y <- c(16, 32, 64)
df <- data.frame(x, y)
df## x y
## 1 a 16
## 2 b 32
## 3 c 64O pacote tibble foi criado como uma atualização12 do data-frame, e pode ser utilizado de maneira similar:
## # A tibble: 3 × 2
## x y
## <chr> <dbl>
## 1 a 16
## 2 b 32
## 3 c 64O data-frame é o formato adequado para armazenar os dados no formato Tidy Data, junto com o tibble (seção 2.8)
2.5.1 Data-frames nativos no R
Existe uma grande quantidade de data-frames pré instalados no R que não necessitam de importação. Alguns dos nossos exemplos irão usar alguns destes data-frames, por exemplo, o data-frame iris, que contém as medidas, em centímetros, do comprimento e largura das pétalas e sépalas de três espécies de plantas: Iris setosa, I. versicolor e I. virginica.
2.6 Importando dados para o R
É muito comum que os dados que utilizaremos em nossas análises estejam armazenadas em fontes externas ao R, como arquivos de texto (*.csv, *.tsv, *.txt), planilhas (*.xlsx, *.ods) ou mesmo grandes bases de dados da internet. A importação consiste na leitura destas fontes para um data-frame no R.
Apesar de existirem vários pacotes e funções para importação de arquivos, vamos apresentar aqueles que fazem parte do tidyverse
2.6.1 Importar arquivos .csv
Os arquivos *.csv (comma-separated values) estão entre os mais populares para distribuição de dados. É um arquivo de texto, cujos valores são delimitador por vírgula.
O pacote readr possui a função read_csv para importação destes arquivos, cujo delimitador entre valores é a vírgula e o separador de decimal é o ponto. Outra função semelhante, read_csv2 importa arquivos .csv cujo delimitador entre valores é o ponto e vírgula e o separador de decimal é a vírgula13, mais comum em alguns países como o Brasil.
O arquivo cbs_jul19.csv (Figura 2.2) contém as temperaturas mínimas e máximas diárias para a cidade de Curitibanos-SC registradas nos primeiros 15 dias do mês de julho de 2019.
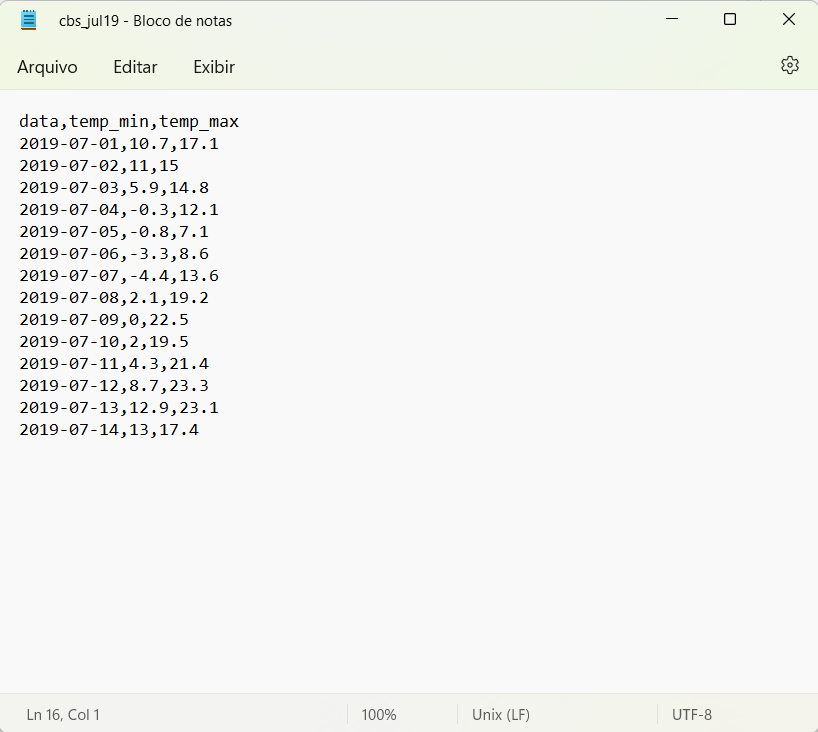
Figura 2.2: Arquivo *.csv cujo delimitador entre valores é a vírgula e o separador de decimal é o ponto.
Os mesmos dados, mas em um arquivo (cbs2_jul19.csv - Figura 2.3) cujo delimitador de valores é o ponto e vírgula e o separador de decimal é a vírgula:
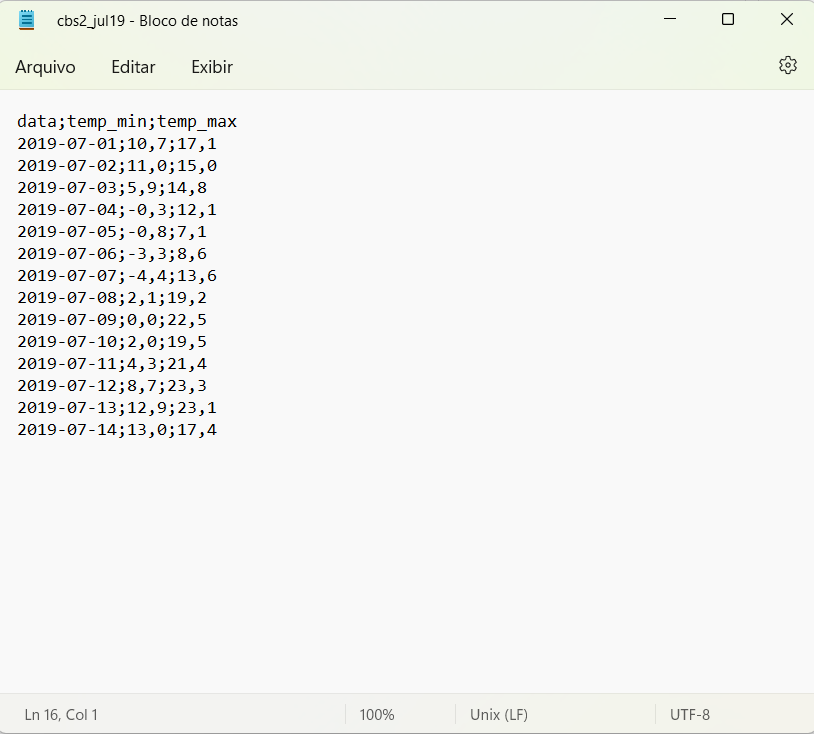
Figura 2.3: Arquivo *.csv cujo delimitador entre valores é o ponto e vírgula e o separador de decimal é a vírgula
A importação com a função read_csv é feita como no exemplo abaixo:
temperaturas <- readr::read_csv("cbs_jul19.csv")
temperaturas## Rows: 14 Columns: 3
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ","
## dbl (2): temp_min, temp_max
## date (1): data
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.## # A tibble: 14 × 3
## data temp_min temp_max
## <date> <dbl> <dbl>
## 1 2019-07-01 10.7 17.1
## 2 2019-07-02 11 15
## 3 2019-07-03 5.9 14.8
## 4 2019-07-04 -0.3 12.1
## 5 2019-07-05 -0.8 7.1
## 6 2019-07-06 -3.3 8.6
## 7 2019-07-07 -4.4 13.6
## 8 2019-07-08 2.1 19.2
## 9 2019-07-09 0 22.5
## 10 2019-07-10 2 19.5
## 11 2019-07-11 4.3 21.4
## 12 2019-07-12 8.7 23.3
## 13 2019-07-13 12.9 23.1
## 14 2019-07-14 13 17.4E com a função csv2:
temperaturas <- readr::read_csv2("cbs2_jul19.csv")
temperaturas## ℹ Using "','" as decimal and "'.'" as grouping mark. Use `read_delim()` for more control.## Rows: 14 Columns: 3
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ";"
## dbl (2): temp_min, temp_max
## date (1): data
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.## # A tibble: 14 × 3
## data temp_min temp_max
## <date> <dbl> <dbl>
## 1 2019-07-01 10.7 17.1
## 2 2019-07-02 11 15
## 3 2019-07-03 5.9 14.8
## 4 2019-07-04 -0.3 12.1
## 5 2019-07-05 -0.8 7.1
## 6 2019-07-06 -3.3 8.6
## 7 2019-07-07 -4.4 13.6
## 8 2019-07-08 2.1 19.2
## 9 2019-07-09 0 22.5
## 10 2019-07-10 2 19.5
## 11 2019-07-11 4.3 21.4
## 12 2019-07-12 8.7 23.3
## 13 2019-07-13 12.9 23.1
## 14 2019-07-14 13 17.42.6.2 Importando arquivos do Excel
Para importar arquivos do Excel, existem a função read_excel do pacote readxl.
O arquivo bflor.xlsx contám dados de um experimento que será estudado no capítulo (10.3).
bflor <- readxl::read_excel("bflor.xlsx")
bflor## # A tibble: 54 × 3
## especie n comprimento
## <chr> <dbl> <dbl>
## 1 Hb 1 47.1
## 2 Hb 2 46.8
## 3 Hb 3 46.8
## 4 Hb 4 47.1
## 5 Hb 5 46.7
## 6 Hb 6 47.4
## 7 Hb 7 46.4
## 8 Hb 8 46.6
## 9 Hb 9 48.1
## 10 Hb 10 48.3
## # ℹ 44 more rows
2.7 O operador pipe: %>%
O operador pipe %>% é uma ferramenta muito útil para expressar uma sequência de ações sobre um objeto. Foi introduzido pela primeira vez no pacote magrittr com o objetivo de facilitar a programação e melhorar e legibilidade do código. Sempre carregue o pacote magrittr antes de usar o pipe.
No exemplo abaixo, as duas linhas de código são equivalentes: aplica-se a função f ao objeto x.
f(x)
x %>% f() # O pipe passa o objeto x para dentro da função fVeja abaixo o pipe em ação:
## [1] 3 4 5 7 10 15 23
x %>% "+"(2) # equivalente à operação anterior## [1] 3 4 5 7 10 15 23## [1] 16 49 100 256 625 1600 4096## [1] 53No caso de uma função com vários argumentos, o pipe passa o objeto para a primeira posição da função, o argumento mais à esquerda:
sample(x, size = 1)## [1] 1## [1] 5No caso de ser necessário que o objeto seja passado para outro argumento à direita, que não seja o primeiro, utilizamos um . como marcador:
iris %>%
lm(Petal.Length ~ Species, data = .) # o objeto iris é passado para o argumento data com um .##
## Call:
## lm(formula = Petal.Length ~ Species, data = .)
##
## Coefficients:
## (Intercept) Speciesversicolor Speciesvirginica
## 1.462 2.798 4.0902.8 Organização dos dados: Tidy data
Para facilitar as análises, é importante que os dados estejam organizados em um formato consistente, chamado de Tidy Data (WICKHAM (2014)). As regras gerais para este formato são:
- Cada variável deve estar em uma coluna;
- Cada observação deve estar em uma linha;
- Cada valor deve ter sua própria célula.
Uma função bastante útil para organizar os dados no fomato tidy é a função tidyr::pivot_longer14.
Considere o conjunto de dados presente nesta planilha do Excel: liquido.xlsx (Figura 2.4)
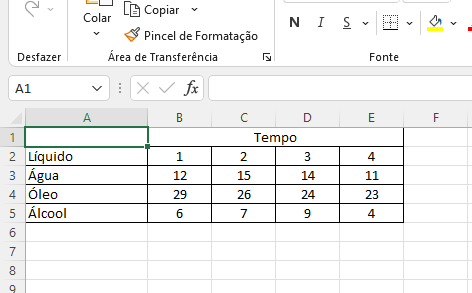
Figura 2.4: Exemplo de dados não organizados no formato tidy em uma planilha do Excel.
Os dados foram digitados de modo que os valores da variável tempo estão espalhados por 4 colunas. A variável repetição (implícita) está em uma linha (cabeçalho das colunas) e o nome das colunas (Tempo) está em uma célula mesclada.
Ao importar este arquivo com a função readxl::read_excel temos como resultado:
messy_data <- readxl::read_excel("liquido.xlsx")
messy_data## New names:
## • `` -> `...1`
## • `` -> `...3`
## • `` -> `...4`
## • `` -> `...5`## # A tibble: 4 × 5
## ...1 Tempo ...3 ...4 ...5
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 Líquido 1 2 3 4
## 2 Água 12 15 14 11
## 3 Óleo 29 26 24 23
## 4 Álcool 6 7 9 4Observamos que a primeira linha (células mescladas) da planilha bagunçou ainda mais o resultado. Vamos importar novamente exlcuindo a primeira linha com o argumento skip = 1.
messy_data <- readxl::read_xlsx("liquido.xlsx", skip = 1)
messy_data## # A tibble: 3 × 5
## Líquido `1` `2` `3` `4`
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 Água 12 15 14 11
## 2 Óleo 29 26 24 23
## 3 Álcool 6 7 9 4Para transformar o data-frame no formato tidy, utilizamos a função tidyr::pivot_longer:
tidy_data <- tidyr::pivot_longer(messy_data, cols = 2:5, names_to = "rep", values_to = "tempo")
tidy_data## # A tibble: 12 × 3
## Líquido rep tempo
## <chr> <chr> <dbl>
## 1 Água 1 12
## 2 Água 2 15
## 3 Água 3 14
## 4 Água 4 11
## 5 Óleo 1 29
## 6 Óleo 2 26
## 7 Óleo 3 24
## 8 Óleo 4 23
## 9 Álcool 1 6
## 10 Álcool 2 7
## 11 Álcool 3 9
## 12 Álcool 4 4-
cols: colunas a serem transformadas para o formato tidy; -
names_to: nome da nova coluna criada a partir das informações armazenadas originalmente nos cabeçalhos das colunas; -
values_to: nome da nova coluna criada a partir dos valores armazenados nas células.
O caminho inverso também pode ser feito, transformando um data-frame do formato longo para o formato wide com a função tidyr::pivot_wider